Ген hgbA имеет особое значение для выживания бактерии Pasteurella multocida в клетке у хозяина и сохранения его патогенности. Ген hgbA может использоваться в качестве маркера в эпидемиологических мониторинговых исследованиях. В ходе экспериментов были наработаны ПЦР продукты гена hgbA, кодирующего железосвязывающие белки пяти бактериальных штаммов Pasteurella multocida, выделенных от сайгаков в Казахстане. Были расшифрованы нуклеотидные последовательности гена hgbA штаммов Pasteurella multocida, выделенных от сайгаков в Казахстане. Нуклеотидная последовательность гена hgbA казахстанского штамма Pasteurella/Saigas/2011/ZKO/KZ была депонирована в Международную базу GenBank под номером KT238895.1. Сравнительный анализ нуклеотидных последовательностей гена hgbA Pasteurella multocida показал, что казахстанские штаммы бактерии Pasteurella multocida, выделенные в 2010, 2011 и 2012 гг. идентичны между собой на 100 %. Нуклеотидные последовательности гена hgbA казахстанских штаммом бактерии Pasteurella multocida показали 99 %-ную гомологию со штаммом Pasteurella multocida_Pm25 (AF331501.1) из Международной базы GenBank. Эти исследования необходимы для выяснения патогенеза и роли гена hgbA в качестве одного из факторов вирулентности бактерии Pasteurella multocida в иммунном процессе.
Введение
Pasteurella multocida — условно-патогенная грамотрицательная бактерия, постоянно обитающая на слизистой оболочке верхних дыхательных путей животных. Бактерия обладает несколькими факторами вирулентности (капсула, дермонекротический токсин, адгезины, протектины, гиалуронидаза, железотранспортирующие протеины и др.), обусловливающими ее патогенность для животных. Геном Pasteurella multocida состоит из одной кольцевой хромосомы длиной в 2257487 пар оснований, содержит приблизительно 2014 кодирующих генов, 6 рибосомальных РНК оперонов и 57 tRNAs [1].
Бактерии производят и высвобождают в окружающую среду микромолекулы с различными химическими структурами, известные как сидерофоры, которые способны хелатировать свободное железо. Сидерофоры как специфические белковые рецепторы участвуют в процессе поглощения железа [2]. И к числу таких сидерофоров относится гемоглобин-связывающий наружный белок А, кодирующийся геном hgbA. Этот ген кодирует белок, который отвечает за регулирование гемоглобина в организме [3].
Роль железа в патогенезе Pasteurella multocida очень важна [4]. Существует два независимых механизма приобретения железа белками Pasteurella multocida. Первый механизм — железосвязывающие белки экспрессируются на наружной мембране бактериальной клетки, взаимодействуя непосредственно с железосвязывающим гликопротеином. Второй механизм — бактериальные белки связываются с гемоглобином и гемоглобиннным комплексом [5].
Ген hgbA имеет особое значение для выживания бактерии Pasteurella multocida в клетке у хозяина и сохранения его патогенности. Поэтому целью данной работы является проведение генетического анализа гена hgbA бактерий Pasteurella multocida, выделенных от павших сайгаков в Казахстане.
Ген hgbA может использоваться в качестве маркера в эпидемиологических мониторинговых исследованиях.
Материалы и методы
В эксперименте были использованы ДНК штаммов бактерии рода Pasteurella, выделенные в Казахстане: Pasteurella/Saigas/2010/ZKO/KZ, Pasteurella/Saigas/2011/ZKO/KZ, Pasteurella/Saigas/2012/ Kostanai/KZ, Сайгачий/88 и Сайгачий/88 (телячий).
Выделение ДНК. Выделение ДНК проведено с помощью коммерческого набора «DNeasy® Blood & Tissue Kit (250)», фирмы Qiagen.
Проведение ПЦР. Реакционная смесь для постановки реакции состояла из следующих компонентов: Master mix объем 25 мкл, 10x ПЦР буфер — 2,5 мкл, dNTP — 1 мкл, MgCl2 — 1 мкл; 10 пмоль F праймер — 1 мкл, 10 пмоль R праймер — 1 мкл, 5 ед. Taq DNA Polymerase — 0,5 мкл; Н2О — 13 мкл, ДНК — 3 мкл. Наработка ПЦР-продуктов проводена в термоциклере GeneAmp PCR System 9700 (Applied Biosystems, США) согласно следующему режиму амплификации: 94 °C — 3 мин, 35 цикл, 94 °C — 20 с, 57 °C — 20 с, 72 °C — 40 с и 72 °C — 7 мин. При постановке ПЦР были использованы олигонуклеотидные праймеры: hgbAs -5/- TCA ACG GCA GAT AAT CAG GG -3/ и hgbAa -5/- GCG GGA ATG CTG AAG ATA AG-3/.
Электрофоретический анализ. ПЦР продукты амплификации ДНК бактерии Pasteurella multocida анализированы в 1,5 % агарозном геле в ТАЕ-буфере при напряжении 80 В/см длины геля, в течение 60 мин с дальнейшей детекцией на трансиллюминаторе Gel ChemiDoc («Bio-Rad» США). Полученные результаты были визуализированы и зарегистрированы с помощью программы «Quantity One».
Определение нуклеотидной последовательности. Секвенирование проведено методом дидезоксисеквенирования с использованием терминирующих дидезоксинуклеотидов (метод Сенгера) на автоматическом 16-капиллярном секвенаторе Genrtic Analyser 3130xl, (Applied Biosystems, USA). Последовательность фрагмента ДНК содержала 2167 пар оснований. Нуклеотидная последовательность гена hgbA казахстанского штамма Pasteurella/Saigas/2011/ZKO/KZ была депонирована в международную базу GenBank под номером KT238895.1.
Результаты исследования и их обсуждение
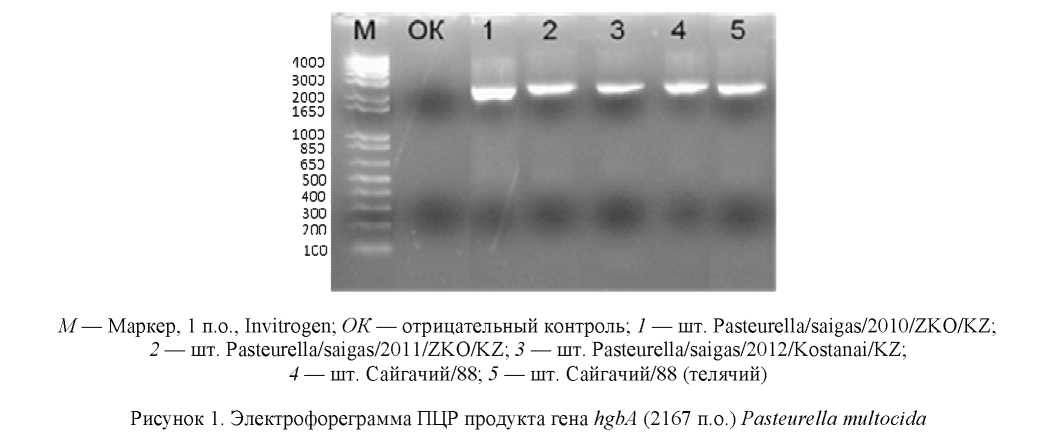
Амплификация гена hgbA проведена в ПЦР с использованием праймеров hgbAs и hgbAa для исследуемого гена. Результаты представлены на рисунке 1.
На рисунке 1 показаны ПЦР фрагменты гена hgbA казахстанских штаммов бактерии Pasteurella multocida с размерами 2167 п.о. Определены нуклеотидные последовательности полученных ПЦР продуктов гена hgbA. Был проведен сравнительный молекулярно-генетический анализ нуклеотидных последовательностей гена hgbA казахстанских штаммов Pasteurella multocida с данными базы GenBank (рис. 2).
|
Description |
Max score |
Total score |
Query cover |
E value |
Ident |
Accession |
|
|
□ |
Pasteurella multocida strain PM25 HgbA gene, complete cds |
3947 |
4720 |
100% |
0.0 |
99% |
AF331501.1 |
|
□ |
Haemophilus influenzae strain NTHi2019 hemoglobin-haptoglobin binding proteiι |
2745 |
3263 |
90% |
0.0 |
92% |
KC607496.1 |
|
□ |
Haemophilus influenzae Rd KW20, complete genome |
2499 |
2499 |
75% |
0.0 |
90% |
L42023.1 |
|
□ |
Pasteurella multocida subsp. multocida str. HB03, complete genome |
1989 |
2702 |
61% |
0.0 |
98% |
CP003328.1 |
|
□ |
Pasteurella multocida 36950, complete genome |
1989 |
2702 |
61% |
0.0 |
98% |
CP003022.1 |
|
□ |
Haemophilus influenzae KR494. complete genome |
1914 |
2432 |
71% |
0.0 |
96% |
CP005967.1 |
|
□ |
Pasteurella multocida putative TonEHdependent receptor IhgbA) gene, complete |
1882 |
2567 |
58% |
0.0 |
98% |
AF237932.1 |
|
□ |
Pasteurella multocida subsp. multocida str. 3480, complete genome |
1847 |
3127 |
88% |
0.0 |
95% |
СР0014Ũ9.1 |
|
□ |
Pasteurella multocida subsp. multocida str. HN06. complete genome |
1847 |
3127 |
88% |
0.0 |
95% |
СР0Ū3313.1 |
|
□ |
Pasteurella multocida subsp. multocida str. Pm70, complete genome |
1831 |
3083 |
88% |
0.0 |
95% |
AE004439.1 |
|
□ |
Actinobacillus actinomvcetemcomitans strain НК10Ŭ2 hemoglobin binding protelι |
193 |
193 |
34% |
1e-44 |
71% |
AF359451.1 |
Рисунок 2. Сравнительный анализ гена hgbA с данными GenBank
Полученные результаты сравнительного анализа показали, что исследуемые казахстанские штаммы по гену hgbA идентичны на 99 % со штаммом Pasteurella multocida_Pm25 (AF331501.1), а также на 98 % со штаммами Pasteurella multocida_HB03 (CP003328.1), Pasteurella multocida_36950 (CP003022.1), Pasteurella multocida_ hgbA gene (AF237932.1).
На рисунке 3 представлены результаты сравнительного анализа казахстанских штаммов между собой и с данными международной базы GenBank по нуклеотидным последовательностям гена hgbA.

По результатам сравнительного анализа нуклеотидных последовательностей гена hgbA видно (рис. 3), что казахстанские штаммы Pasteurella/Saigas/2010/ZKO/KZ, Pasteurella/Saigas/2011/ZKO/KZ, Pasteurella/Saigas/2012/Kostanai/KZ, Сайгачий/88, Сайгачий/88 (телячий) имеют 100 % идентичность между собой. Самым близкородственным к казахстанским образцам оказался штамм Pasteurella multocida_Pm25 (AF331501.1). Pasteurella multocida_Pm25 (AF331501.1) и казахстанские штаммы отличаются от других штаммов из базы GenBank наличием двух нуклеотидных замен: С на Т в позиции 645 и Т на С в позиции 811. Штамм Pasteurella multocida_Pm25 (AF331501.1) имеет одну нуклеотидную замену А на Т в позиции 962 по сравнению со всеми исследуемыми штаммами.
Полученные данные свидетельствуют о том, что ген hgbA может быть использован в качестве биомаркера для выявления вирулентности бактерии Pasteurella multocida.
Заключение
В результате проведенных работ были наработаны ПЦР продукты гена hgbA, кодирующего железосвязывающие белки бактериальных штаммов Pasteurella multocida, выделенных от сайгаков в Казахстане. Расшифрованы нуклеотидные последовательности гена hgbA казахстанских штаммов Pasteurella/Saigas/2010/ZKO/KZ, Pasteurella/Saigas/2011/ZKO/KZ, Pasteurella/Saigas/2012/Kostanai/KZ, «Сайгачий/88», «Сайгачий/88 (телячий)». Проведен сравнительный анализ нуклеотидной последовательности гена hgbA штаммов, выделенных в Казахстане со штаммами Pasteurella multocida из международной базы GenBank. Выявлена тесная родовая связь между штаммами бактерии Pasteurella multocida, выделенных от сайгаков в Казахстане. Также определена 99 %-ная гомология со штаммом Pasteurella multocida_Pm25 (AF331501.1) из международной базы GenBank.
Эти исследования необходимы для выяснения патогенеза и роли гена hgbA в качестве одного из факторов вирулентности бактерии Pasteurella multocida в иммунном процессе.
Список литературы
- May B.J. Complete genomic sequence of Pasteurella multocida, Pm70 / B.J. May, Q. Zhang, L.L. Li, M.L. Paustian, T.S. Whittam, V. Kapur // Proc Natl Acad Sci USA. — 2001. — Р. 3460–3465.
- Boyce J.D. Pasteurella multocida capsule: composition, function and genetics/ J.D. Boyce, J.Y. Chung, B. Adler // Journal of Biotechnology. — 2000. — P. 153–160.
- Ratledge C. Iron metabolism in pathogenic bacteria / C. Ratledge, L.G. Dover // Annu. Rev. Microbiol. — 2000. — Vol. 54. — P. 881–941.
- Tang X. Isolation, antimicrobial resistance, and virulence genes of Pasteurella multocida strains from swine in China / X. Tang, Z. Zhao, J. Hu // Journal of Clinical Microbiology. — 2009. — P. 951–958.
- Cox A.J. Functional characterization of HgbBI a new hemoglobin binding protein of Pasteurella multocida / A.J. Cox, M.L. Hunt, J.D. Boyce, B. Adler // Microb. Pathog. — 2003. — Vol. 34. — P. 287–296.