Антибиотическая резистентность патогенных микроорганизмов на сегодняшний день становится все более опасной проблемой во всем мире, вместе с тем возрастает необходимость в разработке новых антибактериальных мишеней. C момента открытия скользящих зажимов у бактерий было проведено большое количество исследований, в ходе которых открылись его неповторимые свойства — способность связываться с ДНК и увеличивать активность и эффективность белков репараций и репликации, что подчеркивает его большую роль в поддержании устойчивости бактерий к повреждениям ДНК. В настоящий момент количество белков-партнеров, с которыми скользящий зажим способен создавать функциональные комплексы продолжает расти, и потому β-зажим является объектом для внимания как потенциальное решение для поиска новых антибиотиков. В обзорной статье приведены некоторые исследования, освещающие его строение, структуру и механизм действия, а также его способность образовывать комплексы со многими белками-партнерами с помощью уникального мотива связывания β-зажима, который является консервативным и аналогичен для всех белков-партнеров.
Введение
Скользящий зажим — это особый олигомерный белковый комплекс, являющийся субъединицей ДНК-полимераз и имеющий форму кольца молекулярной массой ~82 кДа [1–3]. Они обнаружены практически у всех организмов, включая некоторые вирусы, и называются ядерным антигеном пролиферирующих клеток (PCNA) у эукариот и β-зажимом у прокариот [4]. И хотя во всех этих трех структурах жизни кольцевые зажимы не имеют гомологии в аминокислотных последовательностях, по строению и структуре они практически идентичны. У бактерий скользящий зажим является димером, он состоит из двух β-субъединиц, откуда и получил свое название β-зажим. У архей и эукариот зажимы являются тримером из трёх молекул PCNA. Фаг Т 4 также имеет тримерную структуру зажима и называется gp45 [5].
По своей природе скользящие зажимы являются важнейшим кофактором процессивности полимеразы, увеличивая скорость присоединения нуклеотидов к растущей цепи в тысячи раз. Во время репликации ДНК необходимо точно синтезировать до миллиарда нуклеотидов за короткий период времени, но связь между полимеразой и матрицей достаточно неустойчива и потому репликативные ДНК-полимеразы сами по себе непрерывно синтезируют всего несколько нуклеотидов, прежде чем отпадают из матрицы. Чтобы увеличить степень процессивности для эффективной репликации, ДНК- полимеразы связываются с кольцевыми скользящими зажимами, которые при помощи своей кольцевой формы способны плотно охватить матричную ДНК и крепко удерживать полимеразу, тем самым предотвращая ее диссоциацию от ДНК. Это также указывает на то, что белок — белковое взаимодействие между ДНК, скользящим зажимом и полимеразой более специфично по сравнению с тем, когда ДНК взаимодействует только с полимеразой. Однако, несмотря на это, скользящий зажим, в отличие от других субъединиц, не прикреплен к полимеразе на постоянной основе [4].
Для того, чтобы загрузить замкнутую кольцевую структуру скользящего зажима на ДНК, необходим специальный механизм, способный катализировать временное открытие, сборку и замыкание вокруг ДНК. Такую работу выполняет мультисубъединичный фермент из семейства АТФаз– погрузчик β-зажима (clamp loader). Погрузчик скользящих зажимов (фактор репликации С у эукариот) использует АТФ для размыкания скользящих зажимов и размещения их вокруг 3'-конца матрицы. Помимо этого, погрузчик β-зажимов направляет их на участки инициации синтеза ДНК, а также помогает во взаимодействии β-зажима с ДНК-полимеразами [5].
Не так давно ряд открытий позволил узнать, что, помимо взаимодействия с полимеразой, скользящие зажимы могут участвовать в процессах репликации, модификации и репарации ДНК, связываясь и с другими белками на ДНК (помимо PolS). Опосредует эту связь у белков-партнеров линейный мотив, состоящий из пяти аминокислотных остатков с консенсусной последовательностью QL[SD] LF [1–3].
Универсальный CBM-мотив у прокариот
С β-зажимом образуют комплекс многие репарационные и полимеразные белки, такие как DnaE, PolC, PolIV (DinB), PolV, PolI, MutS, UmuC, DinB1, MutL, XthA, ДНК лигаза и Hda. Все эти белки имеют универсальный β-зажим-связывающий мотив (clamp-binding motif, CBM) с консенсусной последовательностью QL [SD] LF, чаще всего расположенный на С- или N- концах и иногда внутри белка. Данный мотив играет решающую роль в образовании стабильных физических и функциональных связей с β-зажимом. Главным образом, это гидрофобные ароматические аминокислоты, которые связываются с комплементарными остатками, преимущественно на пептид-связывающем кармане (PBG) и реже на С-конце кольцевого зажима [6].
В работе [6] авторы выровняли области, содержащие консенсусные аминокислотные последовательности 250 белков-партнеров кольцевого зажима. Белки-партнеры относились к 6 разным семействам PolB (15 примеров), PolC (22 примера), DnaE1 (72 примера), DinB1 (62 примера), UmuC (20 примеров) и MutS1 (59 примеров) эубактерий, эукариот и архей. Результат показал некоторое сходство между связывающими мотивами эубактериальных и эукариотических систем. Подчеркивается наличие высокого уровня сохранения Gln в первой позиции — данная аминокислота встречалась с частотой 76,4 %, и очень редко заменялась Glu (1,6 %), далее Leu, которая встречалась в 41,6 % во второй позиции, в третьей позиции это были Ser (34 %) и Asp (22,8 %), четвертая и пятая позиции отдавали предпочтение Leu (81,2 %) и Phe (76,8 %). Таким образом, авторы вывели консенсусную последовательность — QL [SD] LF. Примечательно то, что белок репарации ошибочного спаривания MutS1 не содержит совпадений с CBM-мотивом, в то время как его эукариотический аналог способен связыватся с PCNA [6– 8].
В исследовании [9] авторы создавали in vivo и in vitro комплексы между эндонуклеазой MtbXthA и зажимом Mtbβ-Clamp. XthA участвуют в начальной и конечной стадиях эксцизионной репарации оснований бактериальной ДНК (BER). В ходе выравнивания последовательностей MtbXthA с его AP- эндонуклеазными гомологами у других бактерий рядом с сайтом связывания ДНК был установлен мотив 239QLRFPKK245, который соответствовал консенсусной последовательности в работе [6]. Для его подтверждения авторы использовали мутант MtbXthAPIP, который содержал мутацию F242S в предполагаемом мотиве. C помощью флуоресцентного анализа была измерена его аффинность связывания с Mtbβ-Clamp, впоследствии чего авторы наблюдали уменьшение степени сродства между белками. Это подтвердило, что предполагаемый мотив, а именно F242, играет важную роль во взаимодействии между двумя белками, и наличие мутации в данном мотиве нарушает связывание. Кроме того, эксперимент при стандартных условиях реакции со стимуляцией эндонуклеазной и экзонуклеазной активности MtbXthA посредством добавления Mtbβ-Clamp показал, что кольцевой зажим приводит к 75 % расщеплению ДНК субстрата при эндонуклеазной активности, а без кольцевого зажима — MtbXthA расщеплял субстрат на 15 %. При экзонуклеазной активности MtbXthA в комплексе с кольцевым зажимом расщеплял субстрат на 65 %, без кольцевого зажима лишь на 11 %. Еще один проведенный эксперимент в этой работе также показал, что Mtbβ-Clamp способен взаимодействовать с MtbXthA, как через бороздку связывания пептида (PBG), локализированный у Mtbβ-Clamp на субсайте I, так и через С-конец cубсайта III. Так, в присутствии ДНК-субстрата комплекс образовался непосредственно через С-конец, в отсутствие ДНК белки взаимодействовали через PBG, что указывает на то, что у β-Clamp, как и у его эукариотического аналога PCNA есть множественные способы взаимодействия с белками-партнерами [9].
В исследованиях, где каталитическая субъединица ДНК-полимеразы V транслезионного синте- за-UmuC E. coli взаимодействует с β-зажимом, был подтвержден CBM-мотив–357QLNLF361, который ранее был идентифицирован в [6]. UmuC в комплексе с UmuD'2 образуют «склонную к ошибкам» ДНК-полимеразу V, являющуюся важнейшей частью SOS-системы и обладающей сильным мутагенным потенциалом, который, в свою очередь, обеспечивает устойчивость бактерий к антибиотикам. Линейное выравнивание показало, что у PolV предполагаемый мотив находится внутри белка, а не на С-конце. Для его подтверждения авторы инкубировали синтетический меченый пептид, содержащий CBM-мотив–357QLNLF361 с β-зажимом, впоследствии чего образованный комплекс был кристаллизирован и идентифицирован методом гель-фильтрации. Сравнение мотивов связывания кольцевого зажима у Pol II, Pol III, Pol IV c UmuC показало, что UmuC демонстрирует уникальное различие: в отличие от других полимераз, его консервативный Phe-361 не попадает в пептид-связующий карман зажима, но это компенсируется взаимодействием между Asn–359 из UmuC и Arg–152 на поверхности зажима, так как при связывании данные остатки находятся близко друг к другу. Они предполагают, что существующие множественные вариации связывания кольцевого зажима мотивом у разных полимераз опосредуется остатками на поверхности именно кольцевого зажима, и напрямую это связано с присутствием ДНК [10].
В исследованиях взаимодействия β-зажима с ДНК-лигазой у Helicobacter pylori, рядом с С- концом белка был идентифицирован мотив 554QEFIRSLF561, соответствующий консенсусной последовательности. ДНК-лигазы играют одну из ключевых ролей в системах репликаций и репараций ДНК, как у прокариот, так и у эукариот, и, в отличие от эукариотической лигазы, во взаимодействии с PCNA роль кольцевого зажима во взаимодействиях с ДНК-лигазой у бактерий плохо изучена. Для подтверждения существования связи авторы кристаллизировали Hpβ-сlamp с синтетическим пептидом, содержащим предполагаемый мотив 554QEFIRSLF56 из ДНК-лигазы. Результат показал наличие комплекса и то, что он так же, как и большинство белков, опосредуется преимущественно гидрофобными остатками. Авторы отметили важность таких остатков на мотиве, как Ile557, Leu560 и Phe561, подчеркивая, что именно они взаимодействуют с PBG кольцевого зажима. Примечательно то, что ранее аналогичный эксперимент на наличие комплекса MtbLigA-Mtbβ-сlamp белков у M. tuberculosis не нашел подтверждения [11].
Результаты анализа взаимодействия β-зажима и белком негативной регуляции инициации репликации Hda (белок, родственный DnaA) у E. coli подтвердили, что Hda напрямую связывается с β- зажимом через идентифицированный консенсусный CBM-мотив QL [SP] LPL, который ранее был так же идентифицирован в [6]. Выравнивание остатков показало то, что, в отличие от большинства других белков, предполагаемый мотив у белков семейства Hda располагается преимущественно на N- конце. Для того, чтобы доказать является ли выделенный мотив связующим кольцевой зажим, авторы индуцировали мутации на гекспапетиде Hda: заменили QLSLPL на QASAPA в первом мутанте и QLSLPL на QLSAAL во втором мутанте. Результаты показали, что данные замены нарушали способность Hda связываться с кольцевым зажимом. Во втором эксперименте были использованы синтетические пептиды, содержащие мотив, аналогичный гексапептиду мотива Hda. Авторы предполагали, что данные пептиды будут конкурировать за кольцевой зажим с Hda, тем самым нарушая способность последних образовывать комплекс. В результате было достигнуто 50 % ингибирование связывания между Hda и β-зажимом, подтверждающим то, что мотив является β-связующим [12].
PCNAPIP-мотив у эукариот
У эукариот и архей ядерный антиген пролиферирующих клеток (proliferating cell nuclear antigen, PCNA) собирается в гомотримерные кольца, каждый протомер состоит из двух идентичных доменов, соединенных междоменной соединительной петлей (IDCL) [5, 13]. PCNA, как и его аналог у бактерий, взаимодействует с белками-партнерами посредством связывания через PIP-мотив (PCNA– interacting protein motif, PIP-box). У PCNA их несколько, это канонический PIP-мотив — QххНххАА, где Н — это алифатический гидрофобный остаток (чаще всего I, L или M); А — ароматический гидрофобный остаток (F, W или Y); х — любая аминокислота [14, 15]. У некоторых белков репарации и репликации был идентифицирован новый мотив, взаимодействующий с PCNA, который получил название KAbox. Данный мотив встречается реже других и состоит из остатков КА-(A / L / I) — (A / L / Q) — x x — (L / V) [16, 17].
В иcследовании [18] авторы идентифицировали новый альтернативный мотив взаимодействия с PCNA–APIM (R / K- F / W / Y- L / I / V / A-L / I / V / A- K / R). Он содержит до 10 N-концевых аминокислот с высококонсервативным Phe и широко распространен так же, как и классический PIP-мотив, но, в отличие от классического PIP-мотива, который присутствует во многих белках репликации ДНК, APIM чаще встречается в репарационных белках, а также у белков, участвующих в контроле клеточного цикла [18]. Как и классический мотив, APIM связывается с PCNA, через его консервативный пептид-связывающий карман, который находится ниже междоменной соединительной петли (IDCL) [19, 20].
Сам белковый кольцевой зажим PCNA, в отличие от бактериального β-зажима, для взаимодействия с PIP-мотивом имеет три идентичных по структуре и функционированию пептид-связывающих карманов (PBG). Каждый карман находится по одному на каждом мономере белка, и потому PCNA способен одновременно связывать три разных лиганда [21].
Загрузчик зажима
Скользящие зажимы не могут свободно связываться с ДНК из-за своей замкнутой кольцевой формы [1, 22, 23], для этого им требуются АТФ-зависимые комплексы — загрузчики зажимов. Будучи членами AAA+ суперсемейства АТФаз, проявляя моторную и геликазную активность и способность разбирать белковые комплексы, загрузчики зажимов катализируют раскрытие скользящего зажима, загружают их на ДНК и замыкают кольцо вокруг него. У эукариот и архей загрузчик зажимов называется фактор репликации С (RFC), у бактерий — это комплекс γ/τ, а у вирусов — gp44/62 [24, 25]. Обычно AAA+ АТФазы создают гексамерные комплексы, но в комплексе β-зажимом загрузчик зажимов является пентамером. В отсутствие АТФ загрузчики зажима очень слабо взаимодействуют с кольцевыми зажимами, а при связывании АТФ они претерпевают конформационные изменения, которые обеспечивают оптимальное взаимодействие с С-концевой поверхностью зажима и способствуют его последующему раскрытию [26, 27].
Загрузчик бактериального зажима состоит из трех субъединиц — δ, δ′ и γ/τ, которые собираются в гетеропентамер δδ′(γ/τ) [26, 28–31]. Хотя субъединицы γ и τ кодируются одним и тем же геном, однако субъединица τ не участвует при загрузке зажима, и его урезание трансляционным сдвигом рамки считывания дает субъединицу γ. Таким образом, зажимный загрузчик, состоящий из δδ′γ, носит название γ-комплекс. Каждая субъединица γ-комплекса имеет одинаковую архитектуру, состоящую из трех отдельных доменов [26]. Субъединицы всего γ-комплекса связываются с β-зажимом, но δ- субъединица является основной точкой контакта и отвечает за раскрытие кольца. В отсутствие АТФ δ-субъединица спрятана внутри γ-комплекса, и связана с соседней δ'-субъединицей. При связывании АТФ взаимодействие между δ и δ’ нарушается, подставляя δ для связи с С-концевой поверхностью закрытого β-зажима. Далее CBM-мотив на δ-субъединице впоследствии связывается с любым пеп- тид-связывающим карманом на зажиме, дестабилизирует и открывает β-зажимное кольцо. После этого данный комплекс с открытым кольцевым зажимом специфически распознает и связывает ДНК, потом происходит гидролиз АТФ, который возвращает загрузчик зажима в низкоафинное состояние, которое приводит к его отсоединению, оставляя ДНК внутри раскрытого кольца. После отсоединения загрузчика электростатические взаимодействия между положительно заряженной внутренней поверхностью кольца скользящего зажима и отрицательно заряженной ДНК приводят к закрытию скользящего зажима вокруг ДНК [32–35].
Петля междоменного соединителя
β-зажим представляет собой димер, каждый мономер которого состоит из трех глобулярных доменов [1]. Между доменами каждого мономера есть по четыре петли междоменного соединителя (Inter-domain connector loop, IDCL). Димерное сопряжение состоит из связи β-нити имеющих принцип соединения «голова к хвосту», в результате чего образуется кольцевая форма. Область пептид- связующего кармана, взаимодействующего с CBM-мотивом, находится возле IDCL доменов II и III ( см. рис.). Данный карман состоит из двух субсайтов: субсайта I между доменами II и III, глубина ~8 Å×10 Å и 8,5 Å, и в домене III менее глубокого ~ 4,5 Å-субсайта II.
Каноническим мотивом пептидного взаимодействия является QL [SD] LF, где остатки LF связываются с субсайтом I пептид-связывающего кармана на втором домене, в то время как QL ориентированы на субсайт 2 пептид-связывающего кармана в третьем домене [36–39].
На рисунке ниже представлены архитектурные различия в прокариотических и эукариотических ДНК-зажимах, области связывания белка на зажимах. Доменная архитектура скользящих зажимов ДНК: (I) прокариотический мономер β-зажима PCNA состоит из 3 доменов, каждый из которых соединен петлей междоменного коннектора (IDCL). (II) димер бактериального зажима. Каждая субъединица здесь окрашена по-разному. Области взаимодействия с белками на зажиме показаны красным и голубым цветами. Красным отмечен субстайт I, а голубым — субсайт II на C-конце. Вместе они являются пептид-связывающим карманом (PBG). (III) зажим эукариотической ДНК, мономер ядерного антигена пролиферирующих клеток (PCNA), состоит из двух доменов, соединенных IDCL.
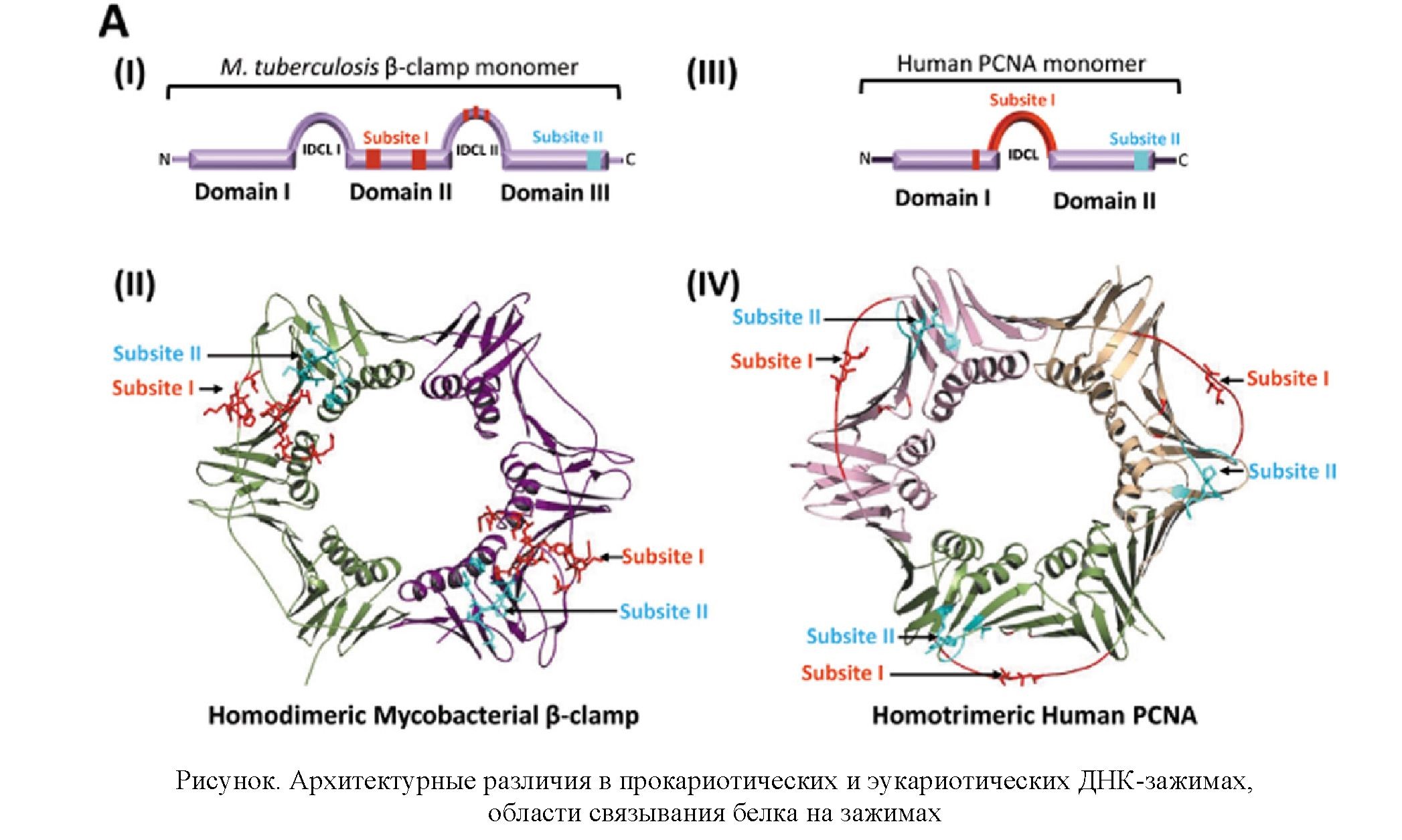
(IV) PCNA человека является тримером. Области, взаимодействующие с белками, также обозначены как субсайт I (красный) и субсайт II (голубой) [9].
Заключение
Бактериальная реплисома является важным объектом для разработки новых антибиотиков для борьбы с лекарственно-устойчивыми штаммами, и бактериальные β-зажимы вызвали большой интерес как потенциальные мишени для антибактериальной терапии, поскольку являются важным компонентом репликативного и репарационного механизма: все пять ДНК-полимераз, включая множество других белков-партнеров, число которых продолжает расти, взаимодействуют с одним и тем же сайтом на зажиме, и, что особенно важно, ингибиторы связывания прокариотических белков не влияют на взаимодействия эукариотических PCNA-связывающих партнеров из-за отсутствия гомологии в последовательностях. Отсюда следует, что поиск новых белков партнеров, способных создавать комплексы с кольцевым зажимом через уникальные пептидные последовательности, — довольно актуальное направление в научной среде. Мы уже увидели, то, что остатки Leu и Phe этих последовательностей являются неотъемлемой частью связывающего взаимодействия. Рациональный дизайн ингибиторов пептид-связывающего кармана на основе таких тонких аспектов связывания мотивом может привести к разработке, как антибиотиков широкого спектра, так и видоспецифичных антибиотиков.
Исследование в первую очередь делает вклад не только для дальнейшего понимания внутриклеточных механизмов бактерий, но и, в частности, для подробного изучения бактериальной репарации и репликации ДНК как ключа для понимания устойчивости патогенов человеческого организма.
Исследование выполнено при финансовой поддержке Комитета науки Министерства образования и науки Республики Казахстан. (Грант № AP08856811).
Список литературы
- Kong, X.P., Onrust, R., O'Donnell, M., & Kuriyan, J. (1992). Three-dimensional structure of the beta subunit of E. coli DNA polymerase III holoenzyme: a sliding DNA clamp. Cell, 69 (3); 425–437. https://doi.org/10.1016/0092–8674(92)90445-i
- Goedken, E.R., Kazmirski, S.L., Bowman, G.D., O'Donnell, M., & Kuriyan, J. (2005). Mapping the interaction of DNA with the Escherichia coli DNA polymerase clamp loader complex. Nature structural & molecular biology, 12(2); 183–190. https://doi.org/10.1038/nsmb889
- Moldovan, G.L., Pfander, B., & Jentsch, S. (2007). PCNA, the maestro of the replication fork. Cell, 129(4); 665–679. https://doi.org/10.1016/j.cell.2007.05.003
- Mizrahi, V., Henrie, R.N., Marlier, J.F., Johnson, K.A., & Benkovic, S. J. (1985). Rate-limiting steps in the DNA polymerase I reaction pathway. Biochemistry, 24(15); 4010–4018. https://doi.org/10.1021/bi00336a031
- Hedglin, M., Kumar, R., & Benkovic, S.J. (2013). Replication clamps and clamp loaders. Cold Spring Harbor perspectives in biology, 5(4); a010165. https://doi.org/10.1101/cshperspect.a010165
- Dalrymple, B.P., Kongsuwan, K., Wijffels, G., Dixon, N.E., & Jennings, P.A. (2001). A universal protein-protein interaction motif in the eubacterial DNA replication and repair systems. Proceedings of the National Academy of Sciences of the United States of America, 98(20); 11627–11632. https://doi.org/10.1073/pnas.191384398
- Johnson, R.E., Kovvali, G.K., Guzder, S.N., Amin, N.S., Holm, C., Habraken, Y., Sung, P., Prakash, L., & Prakash, S. (1996). Evidence for involvement of yeast proliferating cell nuclear antigen in DNA mismatch repair. The Journal of biological chemistry, 271 (45); 27987–27990. https://doi.org/10.1074/jbc.271.45.27987
- Clark, A.B., Valle, F., Drotschmann, K., Gary, R.K., & Kunkel, T.A. (2000). Functional interaction of proliferating cell nuclear antigen with MSH2-MSH6 and MSH2-MSH3 complexes. The Journal of biological chemistry, 275(47); 36498–36501. https://doi.org/10.1074/jbc.C000513200
- Khanam, T., Rai, N., & Ramachandran, R. (2015). Mycobacterium tuberculosis class II apurinic/apyrimidinic- endonuclease/3'-5' exonuclease III exhibits DNA regulated modes of interaction with the sliding DNA β-clamp. Molecular microbiology, 98(1); 46–68. https://doi.org/10.1111/mmi.13102
- Patoli, A.A., Winter, J.A., & Bunting, K.A. (2013). The UmuC subunit of the E. coli DNA polymerase V shows a unique interaction with the β-clamp processivity factor. BMC structural biology, 13; 12. https://doi.org/10.1186/1472–6807–13–12
- Pandey, P., Tarique, K.F., Mazumder, M., Rehman, S.A., Kumari, N., & Gourinath, S. (2016). Structural insight into β-Clamp and its interaction with DNA Ligase in Helicobacter pylori. Scientific reports, 6; 31181. https://doi.org/10.1038/srep31181
- Kurz, M., Dalrymple, B., Wijffels, G., & Kongsuwan, K. (2004). Interaction of the sliding clamp beta-subunit and Hda, a DnaA-related protein. Journal of bacteriology, 186 (11); 3508–3515. https://doi.org/10.1128/JB.186.11.3508-3515.2004
- Matsumiya, S., Ishino, Y., & Morikawa, K. (2001). Crystal structure of an archaeal DNA sliding clamp: proliferating cell nuclear antigen from Pyrococcus furiosus. Protein science: a publication of the Protein Society, 10(1); 17–23. https://doi.org/10.1110/ps.36401
- De Biasio, A., & Blanco, F.J. (2013). Proliferating cell nuclear antigen structure and interactions: too many partners for one dancer? Advances in protein chemistry and structural biology, 91; 1–36. https://doi.org/10.1016/B978-0-12-411637-5.00001-9
- Havens, C.G., & Walter, J.C. (2009). Docking of a specialized PIP Box onto chromatin-bound PCNA creates a degron for the ubiquitin ligase CRL4Cdt2. Molecular cell, 35 (1); 93–104. https://doi.org/10.1016/j.molcel.2009.05.012
- Liang, Z., Diamond, M., Smith, J.A., Schnell, M., & Daniel, R. (2011). Proliferating cell nuclear antigen is required for loading of the SMCX/KMD5C histone demethylase onto chromatin. Epigenetics & chromatin, 4(1); 18. https://doi.org/10.1186/1756- 8935-4-18
- De Biasio, A., Campos-Olivas, R., Sánchez, R., López-Alonso, J.P., Pantoja-Uceda, D., Merino, N., Villate, M., Martin- Garcia, J.M., Castillo, F., Luque, I., & Blanco, F.J. (2012). Proliferating cell nuclear antigen (PCNA) interactions in solution studied by NMR. PloS one, 7(11); e48390. https://doi.org/10.1371/journal.pone.0048390
- Gilljam, K.M., Feyzi, E., Aas, P.A., Sousa, M.M., Müller, R., Vågbø, C.B., Catterall, T.C., Liabakk, N.B., Slupphaug, G., Drabløs, F., Krokan, H.E., & Otterlei, M. (2009). Identification of a novel, widespread, and functionally important PCNA-binding motif. The Journal of cell biology, 186 (5); 645–654. https://doi.org/10.1083/jcb.200903138
- Müller, R., Misund, K., Holien, T., Bachke, S., Gilljam, K.M., Våtsveen, T.K., Rø, T.B., Bellacchio, E., Sundan, A., & Otterlei, M. (2013). Targeting proliferating cell nuclear antigen and its protein interactions induces apoptosis in multiple myeloma cells. PloS one, 8(7); e70430. https://doi.org/10.1371/journal.pone.0070430
- Sebesta, M., Cooper, C., Ariza, A., Carnie, C.J., & Ahel, D. (2017). Structural insights into the function of ZRANB3 in replication stress response. Nature communications,8; 15847. https://doi.org/10.1038/ncomms15847
- Stodola, J.L., & Burgers, P.M. (2016). Resolving individual steps of Okazaki-fragment maturation at a millisecond timescale. Nature structural & molecular biology, 23(5); 402–408. https://doi.org/10.1038/nsmb.3207
- Onrust, R., Stukenberg, P.T., & O'Donnell, M. (1991). Analysis of the ATPase subassembly which initiates processive DNA synthesis by DNA polymerase III holoenzyme. The Journal of biological chemistry, 266(32); 21681–21686.
- Stukenberg, P.T., Studwell-Vaughan, P.S., & O'Donnell, M. (1991). Mechanism of the sliding beta-clamp of DNA polymerase III holoenzyme. The Journal of biological chemistry, 266(17); 11328–11334.
- Neuwald, A.F., Aravind, L., Spouge, J.L., & Koonin, E.V. (1999). AAA+: A class of chaperone-like ATPases associated with the assembly, operation, and disassembly of protein complexes. Genome research, 9 (1); 27–43.
- Erzberger, J.P., & Berger, J.M. (2006). Evolutionary relationships and structural mechanisms of AAA+ proteins. Annual review of biophysics and biomolecular structure, 35; 93–114. https://doi.org/10.1146/annurev.biophys.35.040405.101933
- Bowman, G.D., O'Donnell, M., & Kuriyan, J. (2004). Structural analysis of a eukaryotic sliding DNA clamp-clamp loader complex. Nature, 429 (6993); 724–730. https://doi.org/10.1038/nature02585
- Simonetta, K.R., Kazmirski, S.L., Goedken, E.R., Cantor, A.J., Kelch, B.A., McNally, R., Seyedin, S.N., Makino, D.L., O'Donnell, M., & Kuriyan, J. (2009). The mechanism of ATP-dependent primer-template recognition by a clamp loader complex. Cell, 137(4); 659–671. https://doi.org/10.1016/j.cell.2009.03.044
- Jeruzalmi, D., O'Donnell, M., & Kuriyan, J. (2001). Crystal structure of the processivity clamp loader gamma (gamma) complex of E. coli DNA polymerase III. Cell, 106 (4); 429–441. https://doi.org/10.1016/s0092-8674(01)00463-9
- Oyama, T., Ishino, Y., Cann, I.K., Ishino, S., & Morikawa, K. (2001). Atomic structure of the clamp loader small subunit from Pyrococcus furiosus. Molecular cell, 8 (2); 455–463. https://doi.org/10.1016/s1097-2765(01)00328-8
- Seybert, A., Singleton, M.R., Cook, N., Hall, D.R., & Wigley, D.B. (2006). Communication between subunits within an ar- chaeal clamp-loader complex. The EMBO journal, 25 (10); 2209–2218. https://doi.org/10.1038/sj.emboj.7601093
- Guenther, B., Onrust, R., Sali, A., O'Donnell, M., & Kuriyan, J. (1997). Crystal structure of the delta' subunit of the clamploader complex of E. coli DNA polymerase III. Cell, 91 (3); 335–345. https://doi.org/10.1016/s0092-8674(00)80417-1
- Leu, F.P., Hingorani, M.M., Turner, J., & O'Donnell, M. (2000). The delta subunit of DNA polymerase III holoenzyme serves as a sliding clamp unloader in Escherichia coli. The Journal of biological chemistry, 275(44); 34609–34618. https://doi.org/10.1074/jbc.M005495200
- Turner, J., Hingorani, M.M., Kelman, Z., & O'Donnell, M. (1999). The internal workings of a DNA polymerase clamploading machine. The EMBO journal, 18 (3); 771–783. https://doi.org/10.1093/emboj/18.3.771
- Stewart, J., Hingorani, M.M., Kelman, Z., & O'Donnell, M. (2001). Mechanism of beta clamp opening by the delta subunit of Escherichia coli DNA polymerase III holoenzyme. The Journal of biological chemistry, 276(22); 19182–19189. https://doi.org/10.1074/jbc.M100592200
- Leu, F.P., & O'Donnell, M. (2001). Interplay of clamp loader subunits in opening the beta sliding clamp of Escherichia coli DNA polymerase III holoenzyme. The Journal of biological chemistry, 276 (50); 47185–47194. https://doi.org/10.1074/jbc.M106780200
- Bunting, K.A., Roe, S.M., & Pearl, L.H. (2003). Structural basis for recruitment of translesion DNA polymerase Pol IV/DinB to the beta-clamp. The EMBO journal, 22 (21); 5883–5892. https://doi.org/10.1093/emboj/cdg568
- Burnouf, D.Y., Olieric, V., Wagner, J., Fujii, S., Reinbolt, J., Fuchs, R.P., & Dumas, P. (2004). Structural and biochemical analysis of sliding clamp/ligand interactions suggest a competition between replicative and translesion DNA polymerases. Journal of molecular biology, 335 (5); 1187–1197. https://doi.org/10.1016/j.jmb.2003.11.049
- Georgescu, R.E., Yurieva, O., Kim, S.S., Kuriyan, J., Kong, X.P., & O'Donnell, M. (2008). Structure of a small-molecule inhibitor of a DNA polymerase sliding clamp. Proceedings of the National Academy of Sciences of the United States of America, 105(32); 11116–11121. https://doi.org/10.1073/pnas.0804754105
- Altieri, A.S., & Kelman, Z. (2018). DNA Sliding Clamps as Therapeutic Targets. Frontiers in molecular biosciences, 5; 87. https://doi.org/10.3389/fmolb.2018.00087