В статье представлена характеристика основных полиморфизмов гена рецептора витамина D (VDR): rs2228570 (FokI), rs731236 (TaqI), rs1544410 (BsmI) и rs7975232 (ApaI). Описана роль гормональноактивной формы витамина D (1,25(OH)2D3, кальцитриол) как фактора транскрипции, регулирующего экспрессию генов в клетках-мишенях путем связывания с белком-рецептором витамина D. Отмечено иммуномодулирующее и опосредующее влияние рецепторов VDR на биологические функции организма человека. Дано описание гена рецептора витамина D, и указан его полиморфный характер. Проведен анализ четырех наиболее значимых однонуклеотидных полиморфизмов (SNP) гена VDR. Приведено подробное описание каждого полиморфизма, его геномной позиции, характера взаимодействия с другими полиморфизмами гена рецептора витамина D, а также его влияние на структуру и активность белка VDR. Представлен анализ аллельного состава указанных однонуклеотидных полиморфизмов по литературным источникам и специализированным базам данных SNP. Изучены частота встречаемости отдельных аллелей каждого полиморфизма, а также их влияние на предрасположенность и течение различных заболеваний. Показана необходимость проведения дальнейших исследований полиморфизмов гена VDR, их аллельного состава и распространенности, а также возможностей их потенциального использования в качестве генетических маркеров для таких актуальных, но мало изученных патологий, как COVID–19.
Введение
Витамин D был открыт и долгое время изучался как основной фактор фосфорно-кальциевого обмена, который участвует в формировании и нормальном функционировании костной ткани [1, 2]. Однако дальнейшие исследования показали, что в процессе метаболизма в печени и почках образуется гормонально-активная форма витамина D — 1,25-дигидроксивитамин D3 (1,25(OH)2D3, кальци- триол), роль и возможные эффекты которого гораздо шире [3].
1,25(OH)2D3 является плюрипотентным гормоном, обладающим иммуномодулирующими функциями [4, 5]. Свое эндокринное действие он реализует путем связывания с рецептором витамина D (vitamin D receptor, VDR), который принадлежит к суперсемейству факторов транскрипции, члены которого обладают уникальным свойством непосредственно активироваться небольшими липофильными соединениями [6–9]. Похожим образом функционируют стероидные рецепторы. Рецептор витамина D способен специфически взаимодействовать с 1,25-дигидроксивитамином D3, опосредовать его действие и продуцировать различные эффекты, влияющие на протекание биологических процессов в организме человека [4, 10, 11]. Также VDR функционирует как рецептор литохолевой и вторичной желчной кислоты [12].
Рецептор витамина D вместе 1a-гидроксилазой, которая катализирует последнюю и ключевую стадию синтеза активного 1,25-дигидроксивитамина D3, экспрессируется практически во всех тканях организма [10]. По данным L. Fagerberg и соавт. (2014), проводивших анализ тканеспецифической экспрессии человека методом РНК-секвенирования 27 видов тканей, наибольшая активность VDR была выявлена в тканях тонкой, двенадцатиперстной и толстой кишки, почках, коже, легких и желудка. При этом в тканях мозга, поджелудочной железы, яичников и печени уровень экспрессии гена был минимален [13].
Известны два уровня регуляции рецептора витамина D: генный и негенный. В первом случае рецепторы находятся и функционируют в ядрах клеток-мишеней, во втором — в цитоплазматических мембранах [7, 8]. Реализуясь на генном уровне, VDR проявляет себя как лиганд-индуцибельный фактор транскрипции [12]. При этом 1,25(OH)2D3 и VDR образуют комплекс, в состав которого входят специфические ДНК-связывающие домены, которые должны быть общими с сайтом начала транскрипции первичных генов-мишеней рецептора. Основной функцией данных последовательностей ДНК является контроль транскрипции определенных генов. В результате изменяется последовательность синтезируемой матричной РНК и, как следствие, происходит образование новых форм соответствующих белков, участвующих в регуляции физиологических реакций [4, 9, 14, 15].
Характеристика гена рецептора витамина D
Структура рецептора витамина D закодирована в гене VDR (другие возможные названия: NR1I1; PPP1R163). Ген VDR находится в хромосоме 12, локус 12q13.11 [8, 12, 16]. Количество экзонов в данном гене, по данным разных источников, варьируется от 9 до 11 [6, 17–20]. Другие авторы, такие как Tuoresmaki et al. (2014) указывают, что ген VDR состоит из восьми экзонов и шести альтернативно сплайсированных областей, которые расположены в генетически активных частях, содержащих промоторную область [9]. Однако, по данным NCBI (National Center for Biotechnology Information, Национальный центр биотехнологической информации США), количество кодирующих областей ДНК гена VDR составляет 12 (идентификатор гена: 7421, сборка GRCh38.p13 от 28.02.2019) [12].
Полное секвенирование человеческого генома показало, что многие гены являются полиморфными, т.е. в их интронах и экзонах могут выявляться однонуклеотидные замены пар оснований (single nucleotide polymorphisms, SNPs, снипы). По своей сути SNP являются точковыми мутациями, однако различия в терминологии обусловливаются их частотой встречаемости. Таким образом, мутация считается однонуклеотидным полиморфизмом, если встречается более чем у 1 % населения [21– 23].
SNP могут оказывать существенное влияние на скорость и эффективность транскрипции, стабильность матричной РНК, количество и активность синтезируемого белка, а также, в целом, на уровень экспрессии гена [3, 4, 22]. В настоящее время SNP различных генов используют в качестве биологических маркеров при изучении генетических признаков. Также имеется множество исследований, целью которых является выявление зависимости предрасположенности к какому-либо заболеванию и тяжести его течения от наличия или отсутствия конкретных аллелей в полиморфных генах. Количество аллелей одного SNP может варьировать от двух до четырех, а общая встречаемость и преобладание конкретной аллели во многом зависят от этнической принадлежности исследуемой популяции [5, 8, 11, 22].
Существует несколько баз данных SNP, находящихся в открытом доступе. Среди них dbSNP, SNPedia, MirSNP и др. База dbSNP была разработана и поддерживается Национальным центром биотехнологической информации США (NCBI), она содержит обширную информацию не только о структуре и позиции определенных SNP, но и описывает их клиническое значение, историю, а также встречаемость отдельных аллелей данных полиморфизмов среди различных этнических групп [24, 25].
Ген рецептора витамина D является полиморфным. В более ранних исследованиях были представлены данные о наличии в нем более 470 однонуклеотидных полиморфизмов [17, 18, 21]. Наиболее изученными среди полиморфизмов гена VDR являются rs2228570 (FokI), rs731236 (TaqI), rs1544410 (BsmI) и rs7975232 (ApaI). Второе название SNP обусловлено наличием или отсутствием в них сайтов рестрикции для соответствующих ферментов (FokI, TaqI, BsmI, ApaI) [4, 26, 27]. Существует множество исследований, посвященных выявлению связи данных полиморфизмов с такими заболеваниями, как бронхиальная астма, рак, ревматоидный артрит, туберкулез, сахарный диабет, денге, рассеянный склероз, болезнь Паркинсона и др. [1, 3–6, 17, 18, 28, 29].
Полиморфизм rs2228570 (FokI)
Полиморфизм rs2228570 (FokI) расположен в 5'-кодирующей области 2 экзона гена VDR [3, 11, 16]. Геномная позиция: сhr12:47879112 [18, 30]. Данный SNP является единственным из четырех наиболее значимых SNP гена VDR, который изменяет структуру синтезируемого белка, т.е. является миссенс-мутацией. Кроме того, он не связан ни с одним из других полиморфизмов VDR [6, 18, 26].
В более ранних источниках можно встретить другое название данного полиморфизма — rs10735810 [18]. Однако, по данным NCBI (dbSNP), в период с 2006 по 2010 гг. в rs2228570 были объединены семь SNP, включая rs10735810 (рис. 1) [31].
Рисунок 1. Список полиморф
измов, объединенных в rs2228570 [31]
В литературных источниках полиморфизм rs2228570 преимущественно описывается как генетическая вариация замены нуклеотида Т в стартовом кодоне ATG на аллель С. Таким образом, в гене VDR может находиться либо два стартовых кодона ATG, разделенных шестью нуклеотидами, либо один новый сайт инициации ACG. В результате экспрессии гена носителя аллели Т (АТG) синтезируется белок, состоящий из 427 аминокислот, а в случае генотипа С (ACG) данный белок будет включать на три аминокислоты меньше, т.е. 424 [3, 6, 11, 20, 32, 33]. При этом исследования показали, что короткий вариант белка VDR более активен и обладает повышенной транскрипционной активностью по сравнению с полноразмерным белком [3, 16, 26, 27, 33].
По данным dbSNP (сборка 155 от 9 апреля 2021 г.), полиморфизм rs2228570 может иметь четыре варианта аллелей, т.е. A, C, G и Т. При этом средняя частота встречаемости аллелей по агрегированным данным с выборкой 236272 образцов составляет G = 0,611257, А = 0,388743, T = 0,000000 (рис. 2) [34].
Показанное значение для аллели Т и отсутствие информации по аллели С говорят об их крайне редкой встречаемости по сравнению с А и G.
Также среди ряда исследований, помимо описанного для rs2228570 генотипа С>Т, встречаются аллельные комбинации A>G, А>Т. В них рассматриваются либо общая распространенность аллелей данного SNP среди здорового населения, либо их связь с такими заболеваниями, как денге, бронхиальная астма, гепатит В, болезнь Паркинсона и туберкулез [4, 23, 29, 35–39]. Сводные данные приведены в таблице 1.
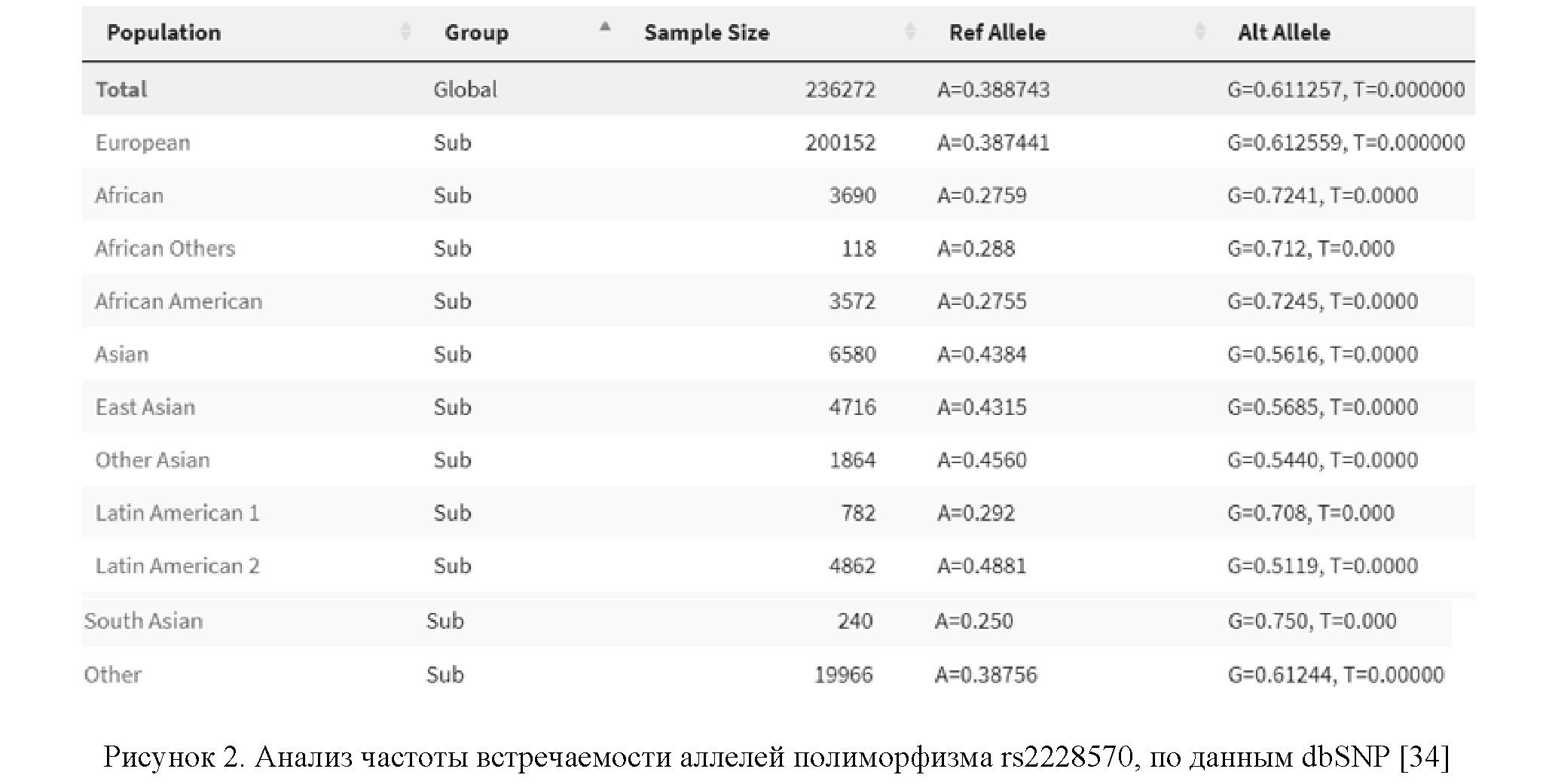
Таблица 1
Анализ изучения аллельных комбинаций rs2228570 в научных публикациях [4, 23, 29, 35–39]
|
Варианты аллелей rs2228570 |
Авторы, год издания публикации |
|
СC / CТ / TT |
Kresfelder et al. (2011), Alagarasu et al. (2012), Grzegorzewska et al. (2014), Bah- rami et al. (2020) |
|
AA / AG / GG |
Osman et al. (2015), Shih-Wei Lee et al. (2016), Hua et al. (2020) |
|
АA /AТ / TT |
Wang et al. (2019) |
Таким образом, ввиду высокой генетической вариабельности, полиморфизм rs2228570 требует дальнейшего изучения распространенности всех его аллелей среди различных этнических групп, а также расширения его роли в качестве генетического маркера заболеваний [22, 29, 35–38].
Полиморфизмы rs731236 (TaqI), rs1544410 (BsmI) и rs7975232 (ApaI)
Полиморфизмы rs731236 (TaqI), rs1544410 (BsmI) и rs7975232 (ApaI) находятся в сильном неравновесном сцеплении друг с другом в 3'-нетранслируемой области гена VDR. Поэтому довольно часто встречается их общее объединенное название: полиморфизм 3' UTR (3' untranslatedregion, 3'-нетранслируемая область) [4, 26, 28, 33]. Данные генетические изменения не влияют на количество аминокислот или их последовательность в белке VDR, но участвуют в регуляции уровня экспрессии гена VDR посредством уменьшения или увеличения стабильности мРНК [3, 11, 16, 27].
rs731236 находится в 9 экзоне гена VDR (геномная позиция chr12: 47844974) [40, 41]. По данным NCBI, в период с 2002 по 2014 гг. в rs731236 были объединены шесть полиморфизмов: rs2228571, rs17777794, rs17880019, rs59730659, rs118037316 и rs386609145 [42]. Согласно данным некоторых авторов [4, 11, 20, 37], этот полиморфизм представляет собой замену аллелей T>C и приводит к молчащим мутациям в соответствующих кодонах. При этом dbSNP (сборка 155 от 9 апреля 2021 г.) характеризует rs731236 как трехаллельный полиморфизм (A, G, T) с преимущественной встречаемостью аллели A=0,61282, меньшей распространенностью G=0,38718 и T=0,000000 (количество образцов в выборке составляет 193424) [43].
Исследования аллелей A и G полиморфизма rs731236 также проводили Osman et al. (2015), S.W. Lee et al. (2016) и Y. Wang et al. (2019). Представлены данные о распространенности данных генетических вариаций среди здорового населения ОАЭ, а также их связь с восприимчивостью к туберкулезу [21, 38, 39].
Полиморфизмы rs1544410 и rs7975232 расположены в интроне 8 [11, 27]. Геномные позиции: chr12: 47846052 и chr12: 47845054 соответственно (рис. 3, 4) [44–46].
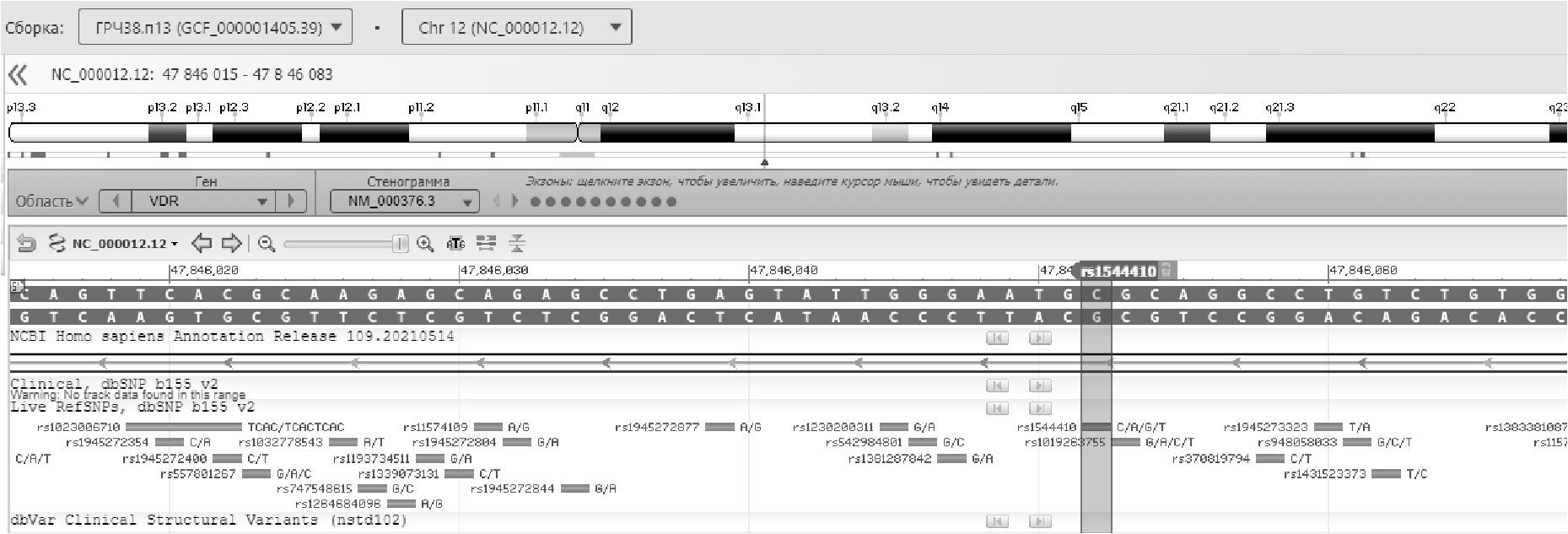
Рисунок 3. Положение rs1544410 в гене VDR [47]
В период с 2008 по 2014 гг. в rs1544410 были объединены три полиморфизма: rs56495123, rs56911380 и rs386536760 [48]. По данным dbSNP, rs1544410 включает в себя все четыре варианта аллелей (A, C, G и Т) [45]. Общая частота встречаемости аллелей в выборке 220674 составила: С=0,611934, Т=0,388066, А=0,000000, G=0,000000 [49]. Таким образом, преобладающими аллелями являются С и Т, однако в литературных данных рассматриваются и другие генетические комбинации (табл. 2).
|
Варианты аллелей rs1544410 |
Авторы, год издания публикации |
|
GG / GA / AA |
Alagarasu et al. (2012), Grzegorzewska et al. (2014), S.W. Lee et al. (2016), P. John et al. (2017) Silva-Ramírez et al. (2018), Yadav et al. (2021) |
|
АA / AТ / TT |
Y. Wang et al. (2019) |
|
GG / GT / TT |
Bahrami et al. (2020) |
rs7975232 был объединен с rs17879735 в 2006 г. [52]. Для данного полиморфизма характерно наличие двух вариантов аллелей: С и А [46]. При анализе общей распространенности среди 39134 образцов преобладающей аллелью является A=0,55448. Встречаемость альтернативной аллели C составляет 0,44552. Однако в различных этнических группах данные показатели могут меняться (рис. 5) [53].
Рисунок 5. Анализ частоты встречаемости аллелей полиморфизма rs7975232, по данным dbSNP [53]
В литературных источниках изучение генотипов СС, СА и АА полиморфизма rs7975232 проводили Alagarasu et al. (2012), S.W. Lee et al. (2016), da Cunha Pereira et al. (2017), Y.Wang et al. (2019), преимущественно в связи с патогенезом туберкулеза [4, 38, 39, 54].
Однако имеется публикация Bahrami (2020) о связи полиморфизмов гена VDR с хроническим гепатитом В, где в качестве аллелей rs7975232 рассматриваются G и T [37].
64
Вестник Карагандинского университета

Заключение
Таким образом, можно сделать вывод, что ген VDR является чрезвычайно полиморфным, и его SNP могут оказывать значительное влияние на протекание различных процессов в организме человека и на его показатели здоровья. Также имеются противоречивые данные об аллельном составе rs2228570 (FokI), rs731236 (TaqI), rs1544410 (BsmI) и rs7975232 (ApaI) среди литературных источников и базой данных dbSNP (NSBI). Одновременно все больше исследований направлено на изучение роли однонуклеотидных полиморфизмов гена VDR в патогенезе различных заболеваний, в числе которых значительное место занимает группа бронхолегочных заболеваний различной этиологии (туберкулез, бронхиальная астма, денге) [3, 4, 8, 12].
Все перечисленное выше делает полиморфизмы гена VDR потенциально значимыми объектами для изучения их влияния на предрасположенность и тяжесть течения других, менее изученных заболеваний. Одним из наиболее актуальных в настоящее время является COVID-19, вызванный коронавирусом SARS-CoV-2 [55].
- Sonomjamts M. Vitamin D plasma concentration and vitamin D receptor genetic variants confer risk of asthma: A comparison study of Taiwanese and Mongolian populations / M. Sonomjamts, H.-F. Kao, Y.-I. Hou, N. Tuvshintur, B. Bayar-Ulzii, N. Logii, J. Yao, L. Shih, H. Wu // World Allergy Organization Journal. — Vol. 12, Iss. 11. — 2019. — P. 210–216. https://doi.org/10.1016/j.waojou.2019.100076
- Пигарова Е.А. Неклассические эффекты витамина D / Е.А. Пигарова, А.А. Петрушкина // Остеопороз и остеопатии. — 2017. — T. 20, № 3. — C. 90–101.
- Zhao D.D. Association of vitamin D receptor gene polymorphisms with susceptibility to childhood asthma: A meta-analysis / D.D. Zhao, D.D. Yu, Q.Q. Ren, B. Dong, F. Zhao, Y.H. Sun // Pediatric Pulmonology. — 2017. — Vol. 52, No. 4. — P. 423–429. https://doi.org/10.1002/ppul.23548
- Kalichamy A. Association of vitamin D receptor gene polymorphisms with clinical outcomes of dengue virus infection / A. Kalichamy, T.P. Honap, A. Mulay, R. Bachal, P.S. Shah, C. Dayaraj // Human Immunology. — 2012. — Vol. 73, Iss. 11. — P. 1194–1199. https://doi.org/10.1016/j.humimm.2012.08.007
- Grant W.B. Evidence that vitamin D supplementation could reduce risk of influenza and COVID-19 infections and deaths / W.B. Grant, H. Lahore, Sh. L. McDonnell, C.A. Baggerly, C.B. French, J.L. Aliano, H.P. Bhattoa // Nutrients. — 2020. — Vol. 12. — P. 98–116. https://doi.org/10.3390/nu12040988
- Hadi S.M. Genotyping of vitamin D receptor FOKI polymorphism as a predictor for type 2 diabetes mellitus by a tetra pri- mer-ARMS-PCR assay / S.M. Hadi, R. Al-Zubaidy // Gene Reports — 2019. — Vol. 15. — P. 1003–10062. https://doi.org/1016/j.genrep.2019.01.003
- Holick M.F. Evaluation, treatment, and prevention of vitamin D deficiency: an Endocrine Society clinical practice guideline / M.F. Holick, N.C. Binkley, H.A. Bischoff-Ferrari, C.M. Gordon, D.A. Hanley, R.P. Heaney, M.H. Murad, C.M. Weaver // J. Clin. Endocrinol. Metab. — 2011. — Vol. 96, No. 7. — P. 1911–1930. https://doi.org/10.1210/jc.2011–0385
- Uitterlinden A.G. Genetics and biology of vitamin D receptor polymorphisms: Review / A.G. Uitterlinden, Y. Fang, J. van Meurs, H.A.P. Pols, J. van Leeuwen // Gene. — 2004. — Vol. 338. — P. 143–156. https://doi.org/10.1015/j.gene.2204.05.014
- Tuoresmäki P. Patterns of Genome-Wide VDR Locations / P. Tuoresmäki, S. Vaisanen, A. Neme, S. Haikkinen, C. Carlberg // PLoS One. — 2014. — Vol. 9, No.4. — P. 96–105. https://doi.org/10.1371/journal.pone.0096105
- Nabih E.S. Association between vitamin D receptor gene FokI polymorphism and atopic childhood bronchial asthma / E.S. Nabih, T.B. Kamel // Egyptian Journal of Chest Diseases and Tuberculosis. — 2014. — Vol. 63, Issue 3. — P. 547–552. https://doi.org/10.1016/j.ejcdt.2014.02.012
- Silva B. Association between Vitamin D receptor gene polymorphisms and pulmonary tuberculosis in a Mexican population / B. Silva, C.A. Saenz-Saenz, L.a. Bracho-Vela, K. Penuelas-Urquides, V. Mata-Tijerina, B.L. Escobedo-Guajardo, N.R. Gonzalez- Rios, O. Vazquez-Monsivais, M.B. de Leon // Indian. J. Tuberc. — 2019. — Vol. 66, No. 1. — P. 70–75. https://doi.org/10.1016/j.ijtb.2018.04.005
- VDR vitamin D receptor [Homo sapiens (human)] [Электронный ресурс]. — Режим доступа:
https://www.ncbi.nlm.nih.gov/gene/7421
- Fagerberg L. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibodybased proteomics / L. Fagerberg, B.M. Hallström, P. Oksvold, C. Kampf, D. Djureinovic, J. Odeberg, M. Habuka, S. Tahmasebpoor, A. Danielsson, K. Edlund, A. Asplund, E. Sjöstedt, E. Lundberg, C.A. Szigyarto // Mol Cell Proteomics. — 2014. — Vol. 13, No. 2. — P. 397–406. https://doi.org/10.1074/mcp.M113.035600
- Carlberg C. Vitamin D receptor signaling mechanisms: Integrated actions of a well-defined transcription factor / C. Carlberg, M.J. Campbell // Steroids. — 2013. — Vol.78, No. 2. — P. 127–136. https://doi.org/10.1016/j.steroids.2012.10.019
- Carlberg C. Dynamics of nuclear receptor target gene regulation / C. Carlberg, S. Seuter // Chromosoma. — 2010. — Vol. 119, No. 5. — P. 479–484. https://doi.org/10.1007/s00412-010-0283-8
- Tabaei S. Vitamin D receptor (VDR) gene polymorphisms and risk of coronary artery disease (CAD): Systematic review and meta-analysis / S. Tabaei, M. Motallebnezhad, S.S. Tabaee // Biochemical Genetics. — 2021. — Vol. 59. — P. 813–836. https://doi.org/10.1007/s10528-021-10038-x
- Tantawy M. Vitamin D receptor gene polymorphism in Egyptian pediatric acute lymphoblastic leukemia correlation with BMD / M. Tantawy, M. Amer, T. Raafat, N. Hamdy // Meta Gene. — 2016. — Vol. 9. — P. 42–46. https://doi.org/10.1016/ j.mgene.2016.03.008
- Mostafaee A. The association analysis between rs1544410 and rs10735810 polymorphisms located at VDR gene and susceptibility to Multiple Sclerosis in Iranian population / A. Mostafaee, S. Rafiei, Z. Fazeli, A. Sayad // Gene Reports. — 2019. — Vol. 17. — P. 1005–1038.
- Taymans S. The human vitamin D receptor gene (VDR) is localized to region 12cen-q12 by fluorescent in situ hybridization and radiation hybrid mapping: Genetic and physical VDR map / S. Taymans, S. Pack, E. Pak, Z. Orban, J. Barsony, Z. Zhuang, C. Stratakis // J Bone Miner Res. — 1999. — Vol. 14, No. 7. — P. 1163–1166. https://doi.org/10.1359/jbmr.1999.14.7.1163
- Yadav U. FokI polymorphism of the vitamin D receptor (VDR) gene and susceptibility to tuberculosis: Evidence through a meta-analysis / U. Yadav, P. Kumar, V. Rai // Infection, Genetics and Evolution. — 2021. — Vol. 92. — P. 1048–1071. https://doi.org/10.1016/j.meegid.2021.104871
- Osman E. Frequency of rs731236 (Taql), rs2228570 (Fok1) of Vitamin-D Receptor (VDR) gene in Emirati healthy population / E. Osman, A. Anouti, G. Elghazali, a. Haq, R. Mirghani, H.A. Sagar // Meta Gene. — 2015. — Vol. 6. — P. 49–52. https://doi.org/10.1016/j.mgene.2015.09.001
- Elkama A. Role of gene polymorphisms in vitamin D metabolism and in multiple sclerosis /A. Elkama, B. Karahalil // ArhHig Rada Toksikol. — 2018. — Vol. 69, No. 1. — P. 25–31. https://doi.org/10.2478/aiht-2018-69-3065
- Bid H.K. Vitamin-D receptor (VDR) gene (Fok-I, Taq-I and Apa-I) polymorphisms in healthy individuals from north Indian population / H.K. Bid, D.K. Mishra, R.D. Mittal // Asian Pac J Cancer Prev. — 2005. — Vol. 6, No. 2. — P. 147–152.
- Cariaso M. SNPedia: a wiki supporting personal genome annotation, interpretation and analysis / M. Cariaso, G. Lennon // Nucleic Acids Research. — 2012. — Vol. 40. — P. 1–5. https://doi.org/10.1093/nar/gkr798
- Sayers E.W. Agarwala R. Database resources of the National Center for Biotechnology Information / E.W. Sayers, J. Beck, E.E. Bolton, D. Bourexis, J.R. Brister, K. Canese, D.C. Comeau, K. Funk, S. Kim, W. Klimke // Nucleic Acids Research. — 2019. – Vol. 47, Issue D1. — P. D23–D28. https://doi.org/10.1093/nar/gkaa892
- Wolski H. Vitamin D receptor gene polymorphisms and haplotypes in the etiology of recurrent miscarriages / H. Wolski, G. Kurzawinska, M. Ozarowski, A. Mrozikiewicz, K. Drews, T.M. Karpinski, A. Bogacz, A. Seremak-Mrozikiewicz // Sci. Rep. — 2021. — Vol. 11. — P. 46–49. https://doi.org/10.1038/s41598-021-84317-3
- Tizaoui K. Association of vitamin D receptor gene polymorphisms with asthma risk: Systematic review and updated metaanalysis of case–control studies / K. Tizaoui, A. Berraies, B. Hamdi, W. Kaabachi, K. Hamzaoui, A. Hamzaoui // Lung. — 2014. — Vol. 192, No. 6. — P. 955–965. https://doi.org/10.1007/s00408-014-9648-8
- John P. Case-control study of vitamin D receptor gene polymorphism in Pakistani rheumatoid arthritis patients / P. John, A. Bhatti, N. Ul Ain, T. Iqbal, T. Sadaf, J.M. Malik // Rev Bras Reumatol. — 2015. — Vol. 57, No. 6. — P. 633–636. https://doi.org/10.1016/j.rbr.2015.01.008
- Hu W. Vitamin D receptor rs2228570 polymorphism and Parkinson’s disease risk in a Chinese population / W. Hu, L. Wang, B. Chen, X. Wang // Neuroscience Letters. — 2020. — Vol. 717. — P. 134–152. https://doi.org/10.1016/j.neulet.2019.134722
- [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/?term=rs2228570
- [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/rs2228570?horizontal_tab=true#history
- Maalmi H. Association of vitamin D receptor gene polymorphisms with susceptibility to asthma in Tunisian children: A case control study / H. Maalmi, F.H. Sassi, A. Berraies, J. Ammar, K. Hamzaoui, A. Hamzaoui // Hum Immunol. — 2013. — Vol. 74, No. 2. — P. 234–240. https://doi.org/10.1016/j.humimm.2012.11.005
- van Etten E. The vitamin D receptor gene FokI polymorphism: Functional impact on the immune system / E. van Etten, L. Verlinden, A. Giulietti, E. Ramos-Lopez // Eur. J. Immunol. — 2007. — Vol. 37, No. 2. — P. 395–405. https://doi.org/10.1002/ eji.200636043
- [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/rs2228570?horizontal_tab=true#frequency_tab
- Kresfelder T.L. Confirmation of an association between single nucleotide polymorphisms in the VDR gene with respiratory syncytial virus related disease in South African children / T.L. Kresfelder, R. Janssen, L. Bont, M. Pretorius, M. Venter // J Med Virol. — 2011. — Vol. 83, No. 10. — P. 1834–1840. https://doi.org/10.1002/jmv.22179
- Grzegorzewska A.E. T-cell cytokine gene polymorphisms and vitamin D pathway gene polymorphisms in end-stage renal disease due to type 2 diabetes mellitus nephropathy: comparisons with health status and other main causes of end-stage renal disease / A.E. Grzegorzewska. G. Ostromecki, P. Zielinska, A. Mostowska, P. Jagodzinski // J Diabetes Res. — 2014. — Vol. 2014. — P. 1203–1217. https://doi.org/10.1155/2014/120317
- Bahrami A. Association of VDR (rs2228570, rs731236, rs7975232, rs1544410) and DBP (rs7041) genes polymorphisms with chronicity of hepatitis B in Iranian patients / A. Bahrami, M. Parsania, A.A. Pourfathollah // Gene Reports. — 2020. — Vol. 19. — P. 1006–1015. https://doi.org/10.1016/j.genrep.2020.100615
- Lee S.W. VDR and VDBP genes polymorphisms associated with susceptibility to tuberculosis in a Han Taiwanese population / S.W. Lee, T.Y. Chuang, H.H. Huang, C. -W. Liu, Y.H. Kao, L.S.H. Wu // J Microbiol Immunol Infect. — 2016. — Vol. 49, No. 5. — P. 783–787. https://doi.org/10.1016/j.jmii.2015.12.008
- Wang Y. A meta-analysis on associations between vitamin D receptor genetic variants and tuberculosis / Y. Wang, H.J. Li // MicrobPathog. — 2019. — Vol. 130. — P. 59–64. https://doi.org/10.1016/j.micpath.2019.02.027
40 Kang S. Vitamin D receptor Taq I polymorphism and the risk of prostate cancer: a meta-analysis / S. Kang, Y. Zhao, L. Wang, J. Liu, X. Chen, X. Liu, Z. Shi, W. Gao, F. Cao // Oncotarget. — 2018. — Vol. 9, No. 6. — P. 7136–7147. https://doi.org/10.18632/oncotarget.23606
41 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/?term=731236
42 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/rs731236?horizontal_tab=true#history
43 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/rs731236?horizontal_tab=true#frequency_tab
44 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/
45 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/?term=rs1544410
46 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/?term=rs7975232
47 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/variation/view/?assm=GCF_000001405.39
48 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/rs1544410?horizontal_tab=true#history
49 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/rs1544410?horizontal_tab=true#frequency_tab
50 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/variation/view/?assm=GCF_000001405.38
51 Selvaraj P. Association of vitamin D receptor gene variants of BsmI, ApaI and FokI polymorphisms with susceptibility or resistance to pulmonary tuberculosis / P. Selvaraj, G. Chandra, S.M. Kurian, A.M. Reetha, P.R. Narayanan // Current Science. — 2003. — Vol. 84, No. 12. — P. 1564–1568.
52 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/rs7975232?horizontal_tab=true#history
53 [Электронный ресурс]. Режим доступа: https://www.ncbi.nlm.nih.gov/snp/rs7975232?horizontal_tab=true#frequency_tab
54 Pereira A. High prevalence of dengue antibodies and the arginine variant of the FcγRIIa polymorphism in asymptomatic individuals in a population of Minas Gerais State, Southeast Brazil / A. Pereira, T.R. de Siquiera, A.A.O. Prado // Immunogenetics. — 2018. — Vol. 70, No. 6. — P. 355–362. https://doi.org/10.1007/s00251-017-1046-y
55 Agshan F.U. Relevance of vitamin D3 in COVID-19 infection / F.U. Afshan, B. Nissar, N.A. Chawdri, B.A. Ganai // Gene Rep. — 2021. — Vol. 24. — P. 1012–1070. https://doi.org/10.1016/j.genrep.2021.101270