Вспышки кори случались примерно каждые 3-5 лет с охватом от 200 до 16 000 человек в год. Казахстанские штаммы вируса кори за этот период представлены 3 генотипами D6/MVi/Berlin.DEU/47.00/, D4/MVi/Bandarabas.IRN/05.10/2/ и D8/MVi/Villupuram.IND/03.07/.
По результатам генотипирования диких штаммов вируса кори, циркулировавших в Республике Казахстан в период 2006 – 2007 гг., была установлена эндемичная трансмиссия штаммов вируса генотипа D6 генетической линии /MVi/Berlin.DEU/47.00/. В 2007 г. циркуляция указанного генотипа была прервана, а очередной подъем заболеваемости, наблюдавшийся в 2010 – 2013 гг., был связан с циркуляцией штаммов вируса кори генотипа D4 генетической линии «MVi/Bandarabas.IRN/05.10/2» Иранского происхождения. В дальнейшем циркуляция штаммов данной генетической линии также была прервана в 2013 году. С начала 2014 года глобальная циркуляция вируса кори характеризуется широким распространением штаммов вируса генотипа D8/MVi/Villupuram.IND/03.07/ Индийского происхождения. Так, штаммы генетической линии «MVi/Villupuram.IND/03.07/[D8]», впервые выделенные в Индии, в 2011 году были импортированы в Таиланд, где вызвали очередную масштабную вспышку кори. Данная вспышка сопровождалась экспортированием инфекции в целый ряд стран, включая нашу Республику.
В этом исследовании мы изучали генетические характеристики диких штаммов вируса кори, циркулировавших на территории Казахстана во время вспышки в 2015 году.
Введение.
Корь продолжает оставаться одной из основных причин детской заболеваемости и смертности в развивающихся странах и угрозой возникновения вспышек в большинстве стран, несмотря на наличие безопасной и эффективной вакцины, используемой более чем 40 лет [1]. В мировом масштабе было достигнуто значительное сокращение смертности от кори, приблизительно с 733,000 смертей в 2000 году до 114 900 в 2014 году [2],[3].
Анализ динамики многолетней заболеваемости корью населения республики Казахстан за 1991-2014 гг. установил цикличность эпидемического процесса, повторяющегося каждые 3-5 лет подъемами и спадом заболеваемости. За наблюдаемый период эпидемия кори наблюдалась с 1993 по 1994 г. В пик этой эпидемии заболеваемость корью населения РК составила 19,52 на 100 тысяч населения. В дальнейшем заболеваемость корью ежегодно снижалась, однако в 1998-2000 гг. вновь была зарегистрирована эпидемия, когда на пике показатель заболеваемости на 100 населения равнялся 12,4. Далее в 2003-2005 гг. имела место эпидемия кори с показателем на пике эпидемии - 106,42 на 100 тысяч населения[4], [5].
Впервые вопрос о ликвидации кори встал в 1975 году, когда под руководством ВОЗ была разработана Расширенная Программа Иммунизации (РПИ), результатом которой, в частности, стало сокращение числа случаев кори на территориях, включившихся в выполнение задачи РПИ. Позднее, в 1991 году, Панамериканская конференция выдвинула задачу элиминации кори на Американском континенте к 2000 г. Результатом усилий, предпринимаемых для достижения поставленной задачи, явилось искоренение кори в США, где с 2000 года не было зарегистрировано ни одного случая этого заболевания, вызванного эндемичными штаммами вируса кори. Та же стратегия намечена в программе Всемирной Организации Здравоохранения «Здоровье для всех», провозгласившей в 1998 году в качестве одной из основных задач XXI века - глобальную ликвидацию кори к 2О20 г. [6].
Для достижения элиминации кори в Казахстане к 2020 году был разработан и реализуется план мероприятий по элиминации кори, краснухи и по профилактике синдрома врожденной краснухи в Республике Казахстан (Приказ МЗ РК №450 от 29.06.2012г.) Одним из важных пунктов плана является молекулярно - генетическая характеристика диких штаммов вируса кори с целью слежения за их циркуляцией среди населения, как важного элемента эпидемиологического надзора. Генотипирование штаммов вируса особенно актуально при достижении фазы элиминации, так как только вирусологический мониторинг позволяет документировать прерывание трансмиссии ранее эндемичных генотипов, что является одним из основных показателей элиминации[7].
Молекулярно - генетическое исследование образцов, выделенных от больных корью в различных регионах, позволяет отслеживать циркуляцию вируса кори и его генетическую изменчивость, выявлять эндемичные штаммы, подтверждать прекращение их циркуляции, дифференцировать завозные случаи, расследовать необычные случаи заболеваний, а также для установления связей, слежения за путем передачи. Недавно созданная база данных «MeaNS» («Measles Nucleotide Surveillance») позволяет ее пользователям обмениваться информацией о сиквенсах и автоматически загружать эти сведения в базы данных ВОЗ.
Для генотипирования ВОЗ рекомендует использовать стандартизированную методику, основанную на анализе нуклеотидной последовательности С - концевого фрагмента N- гена длинной 450 нуклеотидов (наиболее вариабельный участок вирусного генома) и полноразмерного Н - гена. Исследования последних лет, с одной стороны, подтвердили антигенную монотипичность вируса кори, с другой стороны, продемонстрировали генетическую вариабельность диких штаммов. Так, если первая международная классификация генотипов вируса кори ВОЗ (1998) включала 15 генотипов, 11 из которых были активными, то при последнем обновлении в 2005 году (WER, 2005 №7) в нее вошли уже 24 генотипа, из них 18 активных. Большинство генотипов имеют определенное географическое распределение, что позволяет осуществлять мониторинг их циркуляции[8],[9],[10], [11].
Целью настоящего исследования является изучение генетических характеристик диких штаммов вируса кори, циркулировавших на территории Казахстана во время вспышки в 2015 году. Штаммы вируса кори были генотипированы и охарактеризованы на базе Референс лаборатории по контролю за вирусными инфекциями Научно - практического центра санитарноэпидемиологической экспертизы и мониторинга.
Материалы и методы.
Образцы для исследования: Материалом для выделения вируса и вирусной РНК служили образцы мочи, собранные в сроки и по протоколу, рекомендованному ВОЗ, от 18 больных с клиническим диагнозом «корь» и «корь?». Клинический диагноз был подтвержден лабораторно путем выявления специфических коревых IgM в тест-системе иммуноферментного анализа производства компании «Вектор - Бест», Россия.
Выделение вирусной РНК проводили с помощью коммерческого набора Pure Link RNA Mini Kit (Life Technologies, США) согласно инструкции изготовителя.
ПЦР с обратной транскрипцией (ОТ/ПЦР) проводили с использованием праймеров MeV216, MeV214, MeV217, коммерческого набора SuperScript® III One - Step RT-PCR System with Platinum® Taq DNA Polymerase (Life Technologies, США).
Электрофорез ДНК проводили, используя коммерческий комплект реагентов для электрофоретической детекции продуктов амплификации в агарозном геле «ЭФ» - 200 (АмплиСенс, Россия) в 1,7% агарозном геле с трис - боратным буфером концентрированного с бромидом этидия.
Для выделения ДНК из геля использовали коммерческий набор Gel Extraction Kit (Life Technologies, США).
Измерение концентрации очищенной ДНК проводили с использованием коммерческого набора Qubit dsDNA HS (Life Technologies, США).
Секвенирование генома вируса кори осуществляли на автоматическом генетическом анализаторе Genetic Analyzer 3500 (Applied Biosystems, США) с помощью коммерческого набора Big Dye Terminator Kit v3.1 (Applied Biosystems, США). Очистка продуктов секвенирования осуществлялась набором Big Dye Х Terminator Kit (Applied Biosystems, США) в соответствии с инструкцией производителя.
Филогенетический анализ. Нуклеотидные последовательности С - концевого фрагмента N- гена длинной 450 нуклеотидов исследованных образцов сравнивали с последовательностями эталонных штаммов генотипов вируса кори, представленных в базе MeaNS. Анализ нуклеотидных и соответствующих им аминокислотных последовательностей, множественное выравнивание проводили с использованием программного обеспечения MEGA6. Построение филогенетического дерева осуществляли с помощью метода «ближайших соседей» (Neighbor - Joining) программы MEGA6. Статистическую достоверность кластеризации оценивали с использованием метода «множественных повторов» (bootstrap анализ, 500 повторов)[12].
Результаты и обсуждение.
При сравнении нуклеотидных последовательностей 18 проанализированных образцов (образцы РНК) с нуклеотидными последовательностями справочноэталонных штаммов, установлено, что они принадлежат к двум генотипам, при этом большая часть образцов (n=16) относилась к генотипу D8 и 2 образца к генотипу B3 /MVi/Harare.ZWE/38.09./.
По данным филогенетического анализа, генотип D8 представлен 4 доминирующими генетическими линиями, линиями MVi/Villupuram.IND/03.07./D8 (12 обр.), MVs/Republic of Komi.RUS/35.13/D8 (1обр.), MVs/Pretoria.ZAF/19.09./D8 (2обр), MVs/Sohar.OMN/23.12/D8 (1 обр.) ( рисунок 1).
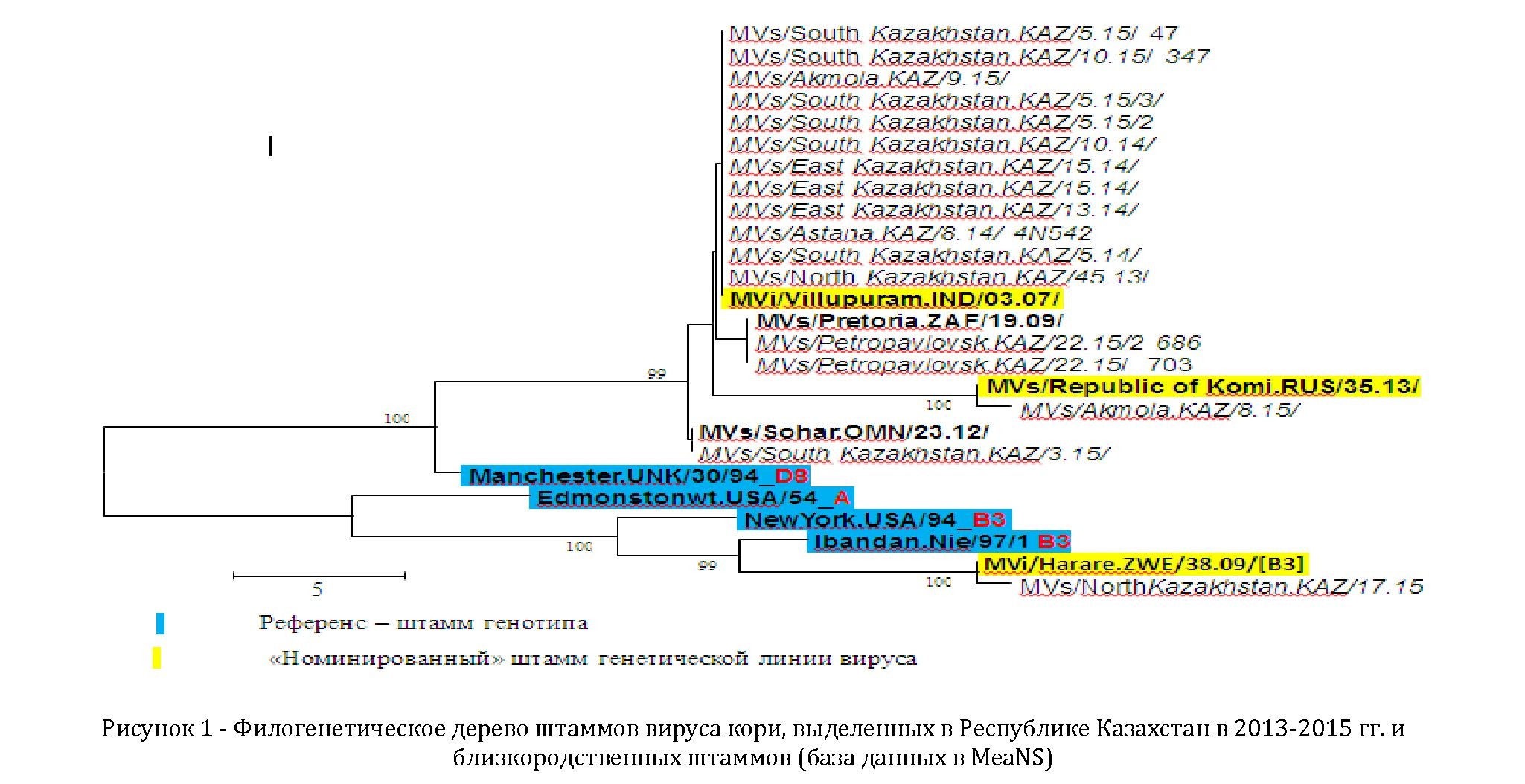 При анализе секвенированных последовательностей штаммов вируса кори за 2015 год, депонированных в международной базе данных MeaNS, нами выявлено, что 12 штаммов вируса кори, выделенных в Северо-Казахстанской (3 обр.), Южно - Казахстанской (7 обр.) и Акмолинской (2 обр.) областях принадлежали к генетической линии «MVi/Villupuram.IND/03.07/[D8]» (Рис.1). Штаммы генетической линии «MVi/Villupuram.IND/03.07/[D8]» впервые выделенные в Индии в 2011г., были импортированы в Таиланд, где вызвали очередную масштабную вспышку кори. Местная циркуляция вируса могла быть обусловлена, в том числе, повторным импортированием из стран, в которых вирусы указанной линии активно циркулировали (Индия, Таиланд, Европейский регион ВОЗ, Россия, Кыргызстан) [13][14], [15], [16]. Еще два штамма, выделенные в 2015 г. в СевероКазахстанской области отличались от штаммов линии «MVi/Villupuram.IND/03.07/[D8]» наличием одной дополнительной нуклеотидной замены и были идентичны штамму «MVs/Pretoria. ZAF/19.09/», выделенному впервые в 2009 г. в Южной Африке, что свидетельствует об их независимом импортировании. Штамм, выделенный в Акмолинской области, близкородственен штаммам генетической линии «MVs/Republic of Komi.RUS/35.13», активно циркулировавшим в Российской Федерации, откуда он, вероятно, был импортирован [17]. Один штамм генотипа D8 «MVs/South Kazakhstan.KAZ/3.15», выделенный в Южно -Казахстанской области, идентичен штамму «MVs/Sohar.OMN/23.12» (Оман, 2012 г.). Так как ранее данный генетический вариант вируса не циркулировал ни в Республике Казахстан, ни в регионе СНГ в целом, позволяет предположить о независимом импортировании инфекции. Кроме того, в 2015 г. впервые в Республике Казахстан был выделен штамм генотипа B3 африканского происхождения в Северо - Казахстанской области. Данный штамм принадлежит к генетической линии «MVi/Harare.ZWE/38.09/[B3]», впервые выделенной в Зимбабве в 2009 г., которая в настоящее время характеризуется широким глобальным распространением с эпизодической изоляцией в большинстве стран [18], [19].
При анализе секвенированных последовательностей штаммов вируса кори за 2015 год, депонированных в международной базе данных MeaNS, нами выявлено, что 12 штаммов вируса кори, выделенных в Северо-Казахстанской (3 обр.), Южно - Казахстанской (7 обр.) и Акмолинской (2 обр.) областях принадлежали к генетической линии «MVi/Villupuram.IND/03.07/[D8]» (Рис.1). Штаммы генетической линии «MVi/Villupuram.IND/03.07/[D8]» впервые выделенные в Индии в 2011г., были импортированы в Таиланд, где вызвали очередную масштабную вспышку кори. Местная циркуляция вируса могла быть обусловлена, в том числе, повторным импортированием из стран, в которых вирусы указанной линии активно циркулировали (Индия, Таиланд, Европейский регион ВОЗ, Россия, Кыргызстан) [13][14], [15], [16]. Еще два штамма, выделенные в 2015 г. в СевероКазахстанской области отличались от штаммов линии «MVi/Villupuram.IND/03.07/[D8]» наличием одной дополнительной нуклеотидной замены и были идентичны штамму «MVs/Pretoria. ZAF/19.09/», выделенному впервые в 2009 г. в Южной Африке, что свидетельствует об их независимом импортировании. Штамм, выделенный в Акмолинской области, близкородственен штаммам генетической линии «MVs/Republic of Komi.RUS/35.13», активно циркулировавшим в Российской Федерации, откуда он, вероятно, был импортирован [17]. Один штамм генотипа D8 «MVs/South Kazakhstan.KAZ/3.15», выделенный в Южно -Казахстанской области, идентичен штамму «MVs/Sohar.OMN/23.12» (Оман, 2012 г.). Так как ранее данный генетический вариант вируса не циркулировал ни в Республике Казахстан, ни в регионе СНГ в целом, позволяет предположить о независимом импортировании инфекции. Кроме того, в 2015 г. впервые в Республике Казахстан был выделен штамм генотипа B3 африканского происхождения в Северо - Казахстанской области. Данный штамм принадлежит к генетической линии «MVi/Harare.ZWE/38.09/[B3]», впервые выделенной в Зимбабве в 2009 г., которая в настоящее время характеризуется широким глобальным распространением с эпизодической изоляцией в большинстве стран [18], [19].
Выводы.
Таким образом, данные молекулярно-генетического мониторинга циркуляции диких штаммов вируса кори за 2015 год демонстрируют трансмиссию в Республике Казахстан штаммов вируса генотипов D8 и B3, ранее на территории страны не циркулировавших. Генетическое разнообразие изолированных штаммов в условиях относительно низкой заболеваемости указывает на преимущественную связь случаев и вспышек инфекции с повторным импортированием вируса. Данные генотипирования свидетельствуют о наличии в республике Казахстан в 2015 году как минимум 5 независимых эпизодов завоза инфекции из-за рубежа из разных источников.
СПИСОК ЛИТЕРАТУРЫ
- Knipe DMH, Peter M. Measles Virus // Fields Virology. - 2007. - Vol. 2. - Р. 1551-1586.
- WHO. Global Measles and Rubella Laboratory Network // MMWR Morb Mortal Wkly Rep. - 2005. - №54. - Р. 1100-1104.
- Global measles mortality // MMWR Morb Mortal Wkly Rep. - 2009. - №58. - Р. 1321-1326.
- Инфекционная заболеваемость в Республике Казахстан за 1991-2014гг. Форма №2 «Отчет об отдельных инфекционных и паразитарных заболеваниях» Научно - практического центра санитарно - эпидемиологической экспертизы и мониторинга;
- Нажмеденова А.Г. Системный подход к анализу эпидемического процесса вакциноуправляемых инфекций. - Алматы: 2010. - 156 с.
- Global Measles and Rubella Strategic Plan 2012-2020 - World Health
- Приказ МЗ РК №450 от 29.06.2012г. «Об утверждении плана мероприятий по элиминации кори и краснухи и профилактике синдрома врожденной краснухи в Республике Казахстан»;
- Mulders MN, Truong AT, Muller CP. Monitoring of measles elimination using molecular epidemiology // Vaccine. - 2001. - №19. - Р. 2245-2249.
- Базаданных Mea NS
- Kremer J.R., Brown K.E., Jin L. et al. High genetic diversity of measles virus. World Health Organization, European region, 2005 - 2006 // Emerg. Infect. Dis. - 2008. - №14(1). - P. 107 - 114.
- Measles virus nomenclature update // Wkly Epidemiol Rec. - 2012. - №87(9). - Р. 73-81.
- Santibanez S., Tischer A., Heider A. et al. Rapid replacement of endemic measles virus genotypes // J. Gen. Virol. - 2002. - №83(11). - P. 2699 - 2708.
- Tamura K, Peterson D, Stecher G, Filipski A, Kumar S. MEGA6: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods // Mol Biol Evol. - 2013. - №30(12). - Р. 2725-2729.
- Rota PA, Bellini WJ. Update on the global distribution of genotypes of wild type measles viruses // J Infect Dis. - 2003. - №187, Suppl 1. - Р. 270-276.
- Rota PA, Brown K, Mankertz A, Santibanez S, Shulga S, Muller CP, Hübschen JM, Siqueira M, Beirnes J, Ahmed H, Triki H, Al-Busaidy S, Dosseh A, Byabamazima C, Smit S, Akoua-Koffi C, Bwogi J, Bukenya H, Wairagkar N, Ramamurty N, Incomserb P, Pattamadilok S, Jee Y, Lim W, Xu W, Komase K, Takeda M, Tran T, Castillo-Solorzano C, Chenoweth P, Brown D, Mulders MN, Bellini WJ, Featherstone D. Global distribution of measles genotypes and measles molecular epidemiology // J Infect Dis. - 2011. - №204, Suppl 1. - Р. 514-523.
- Wairagkar N, Chowdhury D, Vaidya S, Sikchi S, Shaikh N, Hungund L, Tomar RS, Biswas D, Yadav K, Mahanta J, Das VN, Yergolkar P, Gunasekaran P, Raja D, Jadi R, Ramamurty N, Mishra AC, MeaslesNetIndiacollaborators.Molecular epidemiology of measles in India, 2005-2010 // J Infect Dis. - 2011. - №204, Suppl 1. - Р. 403-413.
- Shakya AK, Shukla V, Maan HS, Dhole TN. Identification of different lineages of measles virus strains circulating in Uttar Pradesh, North India // Virol J. - 2012. - №9. - Р. 237-241.
- Heli Harvala, Asa Wiman, Anders Wallensten, Katherina Zakikhany , Helene Englund, Maria Brytting The role of sequencing of the heamagglutinin gene and the hypervariable region in the measles outbreak investigations in Sweden, 2013-2014 // Journal of Infectious Diseases Advance Access. - 2015. - №2. - Р. 48-56.
- Kouomou DW, Nerrienet E, Mfoupouendoun J, Tene G, Whittle H, Wild TF Measles virus strains circulating in Central and West Africa: geographical distribution of two B3 genotypes // J Med Virol. - 2002. - №68. - Р. 433-440.
- Vijesh S. Kuttiatt, Sanughosh Kalpathodi, Sindhu T. Gangadharan, Lalitha Kailas, Easwaran Sreekumar, Suja M. Sukumaran, Radhakrishnan R. Nair.Detection of Measles Virus Genotype B3, India // Emerg Infect Dis. - 2014.- №20(10). - Р. 1764-1766.